Секвенирование древней ДНК
- 1 year ago
- 0
- 0

Бисульфи́тное секвени́рование — общее название группы методов, направленных на изучение паттерна метилирования ДНК посредством обработки её бисульфитом .
Метилирование — первое открытое эпигенетическое маркирование. Оно влияет на уровень экспрессии генов , подавляя транскрипционную активность. Кроме того, во многих случаях метилирование наследуемо , что придает дополнительный интерес его исследованию.
Бисульфит действует на одноцепочечную ДНК , превращая цитозин в урацил . Если же этот цитозин метилирован, то есть к его пятому атому углерода присоединена метильная группа, то такой цитозин не подвергается превращению. Таким образом, бисульфит изменяет последовательность ДНК в зависимости от её паттерна метилирования, и после его воздействия можно установить, какие CpG-динуклеотиды были метилированы, сравнив измененную последовательность с исходной.
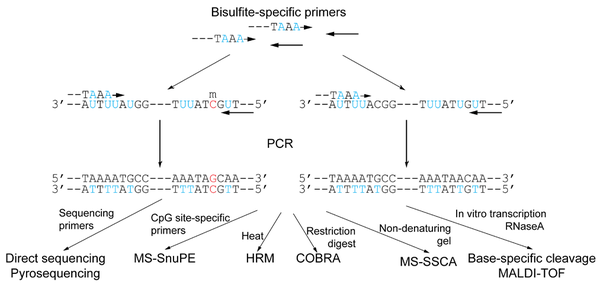
Описанные ниже методы используют секвенирование обработанных бисульфитом участков ДНК для определения паттерна метилирования. Существуют также методы, не основанные на секвенировании, например, комбинированный бисульфитный рестриктационный анализ ( ) и иммунопреципитация метилированной ДНК ( ). Задачи изучения паттернов метилирования могут состоять как в исследовании метилирования конкретного цитозина, так и в определении доли метилированных цитозинов на каком-то участке ДНК, или даже по всему геному в целом. Из приводимых далее методов некоторые больше приспособлены для изучения конкретных сайтов метилирования, другие же — для изучения метилирования на более высоких уровнях. В идеале необходимо определять метилирование каждого аллеля . Описанию методов бисульфитного секвенирования посвящены несколько обзоров .
Первый метод бисульфитного секвенирования описан в 1992 году . Для определения паттерна метилирования использовали ПЦР, праймеры для которой были специфичными как к изменённой, так и к неизменённой бисульфитом ДНК, то есть они не содержали цитозинов, входящих в . Для праймеров использовали участки, близкие к интересующему сайту метилирования, но не содержащие его. В случае, когда цитозин не был метилирован, в амплифицированной последовательности обнаруживался тимин ( урацил ), а в синтезированной комплементарной последовательности — аденин . Если цитозин был метилирован, то в амплифицированной последовательности он и остался, а в комплементарной синтезировался гуанин . Такой метод очень трудозатратен, поскольку требует клонирования продуктов ПЦР для достижения необходимой чувствительности. Эту же проблему можно решить при помощи .
В пиросеквенировании используют ПЦР с праймерами, амплифицирующими как изменённую, так и не изменённую бисульфитом ДНК. Соотношение числа метилированных и неметилированных цитозинов в амплифицируемой последовательности определяется по соотношению количества нуклеотидов А и Г в синтезируемой комплементарной последовательности .
Дальнейшее усовершенствование этого метода заключается в использовании аллель -специфических праймеров с однонуклеотидными полиморфизмами , позволяющих исследовать паттерны метилирования по отдельности в отцовских и материнских аллелях, что особенно полезно при изучении геномного импринтинга .
Этот метод основан на методе анализа ( англ. single-strand conformation polymorphism analysis, SSCA ). SSCA был разработан для выявления однонуклеотидных полиморфизмов (SNP) . Фрагменты ДНК одинаковой длины, но разной нуклеотидной последовательности, с разной скоростью перемещаются в электрофорезе . Вообще говоря, SSCA недостаточно чувствителен для выявления единичной замены, но при достаточном количестве полиморфизмов неметилированных CpG-динуклеотидов в обработанной и необработанной бисульфитом ДНК будет достаточно много, чтобы чувствительности метода оказалось достаточно для определения доли метилированных цитозинов на рассматриваемом участке. Данный метод не позволяет исследовать метилированию в конкретном сайте, однако дает представление о метилировании на уровне некоторого участка или даже всего генома.
Метод (HRM) основан на ПЦР в реальном времени . Температуру повышают с 55 до 95 °C, в результате чего комплементарные связи между цепочками разрушаются. Скорость плавления регистрируют при помощи специальных флуоресцентных красителей, и, зная зависимость флуоресценции от нуклеотидной последовательности цепочек, можно различитьоднонуклеотидные полиморфизмы. Метод не дает информации о том, какие именно CpG-динуклеотиды были метилированы и потому не изменились в процессе бисульфитной обработки, однако дает достаточно точную информацию об их количестве на интересующем участке.
Метод однонуклеотидного расширения праймера исходно был разработан для анализа однонуклеотидных полиморфизмов . Применительно к анализу метилирования его используют следующим образом. На обработанной бисульфитом одноцепочечной ДНК отжигают праймеры до пары оснований, непосредственно предшествующей интересующему сайту метилирования. Затем праймер удлиняетсяпри помощи ДНК-полимеразы с использованием терминирующих её дидезоксинуклеотидов . Соотношение числа метилированных и неметилированных цитозинов в исходной последовательности определяется по соотношению количеств расширений праймеров нуклеотидами Г и А соответственно. Определить соотношение числа метилированных и неметилированных цитозинов в исходной последовательности можно разными способами. Используют радиоактивные или , а также пиросеквенирование .
Кроме того, это можно делать при помощи матрично-активированной лазерной десорбции/ионизации с последующим применением время-пролетного масс-анализатора (MALDI-TOF) или ион-парной обращённо-фазной высокоэффективной жидкостной хроматографии (IP-RP-HPLC) .
Метод основан на использовании специфического расщепления РНК . При добавлении к праймеру в ПЦР промотора РНК-полимеразы in vitro синтезируется РНК-транскрипт интересующего участка, после чего расщепляется рибонуклеазой А . Рибонуклеаза А расщепляет одноцепочечную РНК на местах расположения нуклеотидов Ц и У . Используя устойчивые к расщеплению нуклеозидтрифосфаты dUTP и dCTP, можно добиться специфического расщепления в местах расположения Ц или У. Фрагменты РНК затем анализируют при помощи MALDI - TOF . Данный метод даёт информацию о метилировании каждого из имеющихся CpG-динуклеотидов, а не только о доле метилированных нуклеотидов.
Этот метод использует праймеры, специфичные или только к преобразованной бисульфитом ДНК, или только к непреобразованной . Соответственно, к первым праймерам прикрепляются только участки, которые были метилированы, а ко вторым — те, которые не были, что лишает необходимости секвенировать участки после амплификации.
Амплифицированную в MSP ДНК можно далее анализировать разными другими методами. Метод MethyLight использует MSP в реальном времени , основанную на флуоресценции] . Mc-MSP использует . Высокочувствительный метод плавления использует и то, и другое, и потому обладает достаточной чувствительностью для определения метилирования низкого уровня .
Использование ДНК-микрочипов дает возможность расширить описанные выше методы для анализа метилированности на уровне всего генома . Олигонуклеотиды ДНК-микрочипа делают специфичными к метилированию и соответствующими интересующим CpG-сайтам. Одни комплементарны измененной бисульфитом последовательности (то есть той, в которой CpG-сайт не был метилирован), другие — неизмененной. Примером такого метода служит .
Методы бисульфитного секвенирования имеют ряд ограничений. У млекопитающих широко распространена модификация ДНК 5-гидроксиметилцитозин. При обработке бисульфитом 5-гидроксиметилцитозин превращается в цитозин-5-метилсульфонат, который при секвенировании опознаётся как Ц. Таким образом, бисульфитное секвенирование не может различить 5-гидроксиметилцитозин и , а потому не может рассматриваться как метод, чувствительный только к метилированию ДНК. В 2012 году был разработан метод бисульфитного секвенирования, позволяющий различить эти две модификации .
Поскольку неполное превращение азотистых оснований в ДНК под действием бисульфита, которое возможно только в одноцепочечной ДНК, даёт неправильные результаты, необходима точная подборка условий эксперимента ( температура , концентрация солей), чтобы поддерживать ДНК в денатурированном состоянии . Поддержанию одноцепочечной конформации ДНК может способствовать её закрепление в агарозном геле .
Ещё одна проблема состоит в деградации ДНК при обработке бисульфитом. Поскольку бисульфит действует только на одноцепочечную ДНК, необходимо проводить реакцию в таких условиях, в которых ДНК оставалась бы одноцепочечной, причем проводить её достаточное для успешной конвертации время. Однако, такие условия, а именно высокая температура и длительный период инкубации, могут привести к деградации до 90 % всей ДНК .