Эдвардс против Агиллара
- 1 year ago
- 0
- 0
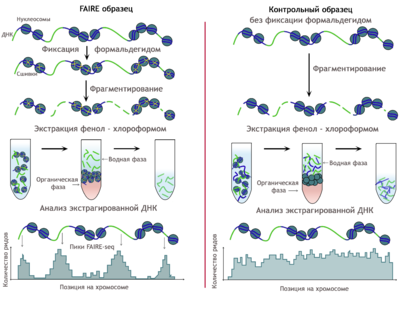
FAIRE-Seq ( англ. Formaldehyde-Assisted Isolation of Regulatory Elements sequencing ) — метод молекулярной биологии , используемый для определения ДНК . FAIRE-Seq является комбинацией метода выделения регуляторных элементов хроматина (FAIRE) и высокопроизводительного секвенирования .

FAIRE — один из методов картирования областей открытого хроматина , наряду с методом определения участков, гиперчувствительных к ДНКазе I ( DNase-seq ) и методом иммунопреципитации хроматина ( ChIP-seq , ChIP-on-chip). Идея метода была предложена в 2003 г. для фракционирования хроматина Saccharomyces cerevisiae на 2 части: первая — участки, транскрибируемые РНК-полимеразой II, вторая — некодирующие последовательности и участки, транскрибируемые РНК-полимеразами I или III . Разделение осуществляется за счет разной степени сшивания формальдегидом отдельных регионов хроматина. В 2007 г. с помощью данного метода были определены регионы, связанные с регуляторной активностью в хроматине человека; таким образом, метод прошел проверку и получил своё название FAIRE . В первоначальном варианте для анализа данных использовалась гибридизация флуоресцентномеченных фрагментов ДНК с ДНК-микрочипами , что в дальнейшем получило название FAIRE-chip. В последующих работах были предложены другие способы анализа полученных участков ДНК, среди которых самым распространённым является FAIRE-seq .
На сегодняшний день существует несколько вариантов метода, предназначенных для тканей животных и растений. При работе с растениями требуются более жесткие условия и продолжительное время фиксации формальдегидом из-за клеточной стенки, восковой кутикулы листьев и воздушных пространств в губчатом мезофилле . Кроме картирования регуляторных участков, метод позволяет сравнивать активные элементы в разных тканях (как опухолевых, так и здоровых). Эксперименты с использованием FAIRE-seq занимают в среднем около трех дней, не включая анализ и интерпретацию полученных данных .
В эукариотических клетках открытый хроматин наблюдается в активных регуляторных областях, поэтому выделение участков, небогатых нуклеосомами, позволило бы картировать регуляторные элементы генома вне зависимости от типа клеток . При добавлении формальдегида к культуре ткани образуются сшивки ( англ. cross-links ) между белками, белками и нуклеиновыми кислотами, в частности ДНК и гистонами или между гистонами. Далее геномная ДНК подвергается ультразвуковой фрагментации на участки длиной в среднем 300—400 пар оснований. При последующей экстракции фенол-хлороформом свободная от белков ДНК будет находиться в водной фазе, а ковалентно связанные ДНК-белковые комплексы — на границе раздела фаз. Таким образом, участки генома, ассоциированные с регуляторной активностью и свободные от нуклеосом (или с небольшим их количеством), находятся над границей раздела фаз в воде. Они могут быть извлечены оттуда, ещё раз очищены от оставшихся белков с помощью фенол-хлороформа, обработаны рибонуклеазой А и переосаждены для последующего анализа. В качестве контроля используется образец ДНК, полученный из той же культуры ткани с помощью тех же процедур, но без фиксации формальдегидом .

Далее в зависимости от метода анализа полученных участков ДНК выделяют FAIRE-seq (высокопроизводительное секвенирование фрагментов ДНК, например используя Illumina ), FAIRE-chip (гибридизация флуоресцентномеченных фрагметов ДНК с ДНК-микрочипами ), FAIRE-qPCR (количественный анализ участков ДНК с помощью ПЦР в реальном времени ). Сейчас FAIRE-seq почти полностью вытеснил другие способы определения экстрагированных фрагментов ДНК .
Для анализа данных секвенирования требуется выравнивать последовательности с референсным геномом, для чего используется, например, Bowtie . Затем производится поиск значимых участков ( significant enrichment regions ). Для этих целей рекомендуется использование ZINBA. В отличие от FAIRE-chip и FAIRE-qPCR, при использовании FAIRE-seq с достаточной глубиной покрытия генома контрольный образец может отсутствовать .
С помощью FAIRE-seq можно осуществлять поиск целевых промоторов опухолевых факторов, обнаружение и проверку потенциальных участков свободного, не связанного с нуклеосомами, хроматина, идентификацию индуцируемых регуляторных участков генома и описание активных регуляторных элементов генома. Метод также позволяет оценивать вариабельность нуклеотидного состава регуляторных участков в различных популяциях .
Применение FAIRE-seq совместно с другими методами, например с DNase-seq, дает более надежный и качественный результат .
Для метода FAIRE не требуется предварительной обработки клеток, так как формальдегид добавляется в питательную среду или напрямую к ткани, быстро приводит к клеточной смерти, что позволяет получить хроматин в состоянии, предшествующем фиксации. При этом разное время фиксации формальдегидом при постоянной концентрации дает одинаковый результат. В отличие от ChIP , метод не зависит от надежности и вариабельности антител. Кроме того, FAIRE не требует использования ферментов, таких как DNase или MNase, которые обычно используются в аналогичных методах для обнаружения областей, свободных от нуклеосом. Отсутствие ферментативной обработки делает этот метод менее вариабельным, более надежным и воспроизводимым .
FAIRE легко адаптируется для использования на образцах тканей, потому что FAIRE не требует одноклеточной суспензии или ядерной изоляции. В этом случае единственным дополнительным шагом является измельчение замороженной ткани в крупный порошок перед фиксацией .
С помощью FAIRE можно выявлять дистальные регуляторные элементы, расположенные далеко от промотеров (например, транскрипционные энхансеры), которые не находятся DNase-seq .
Несмотря на простоту экспериментальной части, для FAIRE-seq, как и для других методов, связанных с обширным секвенированием, необходимо значительное количество вычислительных ресурсов и памяти для интерпретации результатов. В этом плане FAIRE-chip и FAIRE-qPCR могут быть привлекательнее .
По сравнению с DNase-seq и ChIP-seq , FAIRE имеет более низкое отношение сигнал/шум. Поэтому сайты, обнаруженные FAIRE, время от времени могут лишь незначительно отличаться от фонового сигнала, что снижает доверие к идентифицированным сайтам. Этот эффект усугубляется при использовании методов обнаружения, не связанных с секвенированием. Несмотря на низкое отношение сигнал/шум, стоит отметить, что результаты FAIRE имеют хорошую воспроизводимость . Однако обнаружение некоторых промотеров активно экспрессируемых генов осуществляется хуже, чем другими методами, такими как DNase-seq .
Эффективность фиксации тканей варьирует в зависимости от её свойств (чистота, проницаемость, плотность клеток, содержание жира и площадь поверхности), что может затруднять сравнение результатов, полученных для разных тканей .
Данные по геному человека, полученные при помощи FAIRE-seq, доступны как часть проекта ENCODE (энциклопедия элементов ДНК) в интерактивном веб-сайте , который предлагает доступ к данным о геномах различных видов позвоночных , беспозвоночных и крупных модельных организмах , интегрированных с большой коллекцией согласованных аннотаций .