Interested Article - Cas9
- 2020-03-21
- 1
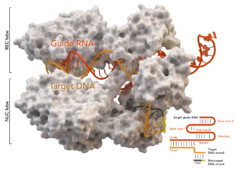

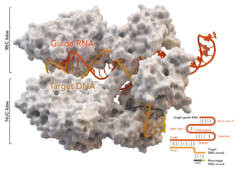
Cas9 ( англ. C RISPR as sociated protein 9 , CRISPR-ассоциированный белок) — это управляемая при помощи РНК -гидов эндонуклеаза , связанная с адаптивной иммунной системой CRISPR ( англ. Clustered Regularly Interspaced Palindromic Repeats ) у ряда бактерий, в частности Streptococcus pyogenes . S. pyogenes использует Cas9 для запоминания , последующей проверки и разрезания чужеродной ДНК , например, ДНК бактериофагов или плазмид .
Cas9 выполняет проверку посредством раскручивания инородной ДНК и определения её комплементарности с двадцатью спаренными основаниями спейсера управляющей РНК . Если субстрат комплементарен управляющей РНК, Cas9 расщепляет чужую ДНК. В этом смысле механизм CRISPR-Cas9 имеет ряд параллелей с механизмом РНК-интерференции (RNAi) у эукариот. Безопасность практического применения данного метода определяется в том числе и тем фактом, является ли искомая последовательность двадцати спаренных оснований уникальной в модифицируемой ДНК.
Использование Cas9 в генной инженерии
Кроме изначальной функции в бактериальном иммунитете, белок Cas9 активно используют для создания точечных разрывов в двойной спирали ДНК, такие разрывы могут приводить к инактивации генов или созданию гетерологичных генов посредством соединения негомологичных концов и соответствующей гомологичной рекомбинации. Вместе с белками и , Cas9 становится значимым инструментом редактирования генома.
Одним из первых продемонстрировал программируемое расщепление ДНК белком Cas9 литовский биохимик Виргиниюс Шикшнис , но его статья не была принята на рассмотрение журналом 18 апреля 2012 года. Месяц спустя он отправил ее в PNAS где на рассмотрение и публикацию ушло несколько месяцев . Тем временем американский биохимик Дженнифер Даудна и французский микробиолог Эмманюэль Шарпантье опубликовали свою статью в рецензируемый научный журнал Science , где она была рассмотрена и принята в течение двух недель .
За создание новых технологий, позволяющих проводить с помощью CRISPR-Cas9 редактирование генома Эмманюэль Шарпантье и Дженнифер Даудна получили Нобелевскую премию по химии 2020 года.
Вызывает удивление тот факт, что из-за патентных ограничений технология редактирования с помощью Cas9 доступна не ученым всего мира, а только владельцам патента, что является препятствием прогрессу науки, разработке лекарств для людей, живущих с серьезными заболеваниями .
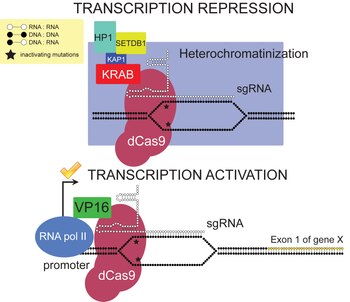
К 2012 году Cas9 приобрёл популярность, потому что он позволяет расщеплять практически любую нуклеотидную последовательность, комплементарную управляющей РНК . Поскольку избирательность Cas9 является следствием комплементарности управляющей РНК и ДНК, а не модификации самого белка (в отличие от случаев TALEN и ZFN), для новых ДНК-мишеней возможна выработка специфических Cas9 . Варианты Cas9, которые связывают ДНК, но не расщепляют её (dCas9), могут быть использованы для доставки транскрипционных активаторов или репрессоров к специфичным последовательностям ДНК с целью регулирования транскрипционной активации и репрессии . Хотя природный Cas9 требует составления управляющей РНК из двух в корне различных РНК — CRISPR-РНК (crRNA), и транс-активационную РНК (tracrRNA) , нацеливание Cas9 было упрощено при помощи выработки единой химерной управляющей РНК. Предполагается, что Cas9 можно будет использовать для изменения генома целых популяций организмов . В 2015 году посредством Cas9 впервые был модифицирован геном человеческого эмбриона . Разработана технология иммуногеномной инженерии гибридом , позволяющая быстро перепрограммировать специфичность их антител с помощью Cas9 .
Создана технология, которая позволяет редактировать отдельные «буквы» ДНК и РНК, не разрезая цепь ДНК, а путём преобразования одного нуклеотидного основания в другое , что позволит лечить врожденные заболевания, вызванные точечными мутациями .
Используя цитидин - деаминазы или же аденозин -деаминазы соединенные с dCas9 можно выключить экспрессию (путем введения преждевременных стоп-кодонов ) или поменять сплайсинг необходимые для синтеза тех или иных белков
Создана технология MAGESTIC (multiplexed accurate genome editing with short, trackable, integrated cellular barcodes), которая не только расщепляет ДНК, но ещё и доставляет к месту разрыва кусок ДНК необходимый для точной замены (с помощью гибридного связывающего ДНК белка -Fkh1p), что повышает точность и эффективность редактирования . Еще одним инструментом для целевой вставки ДНК может стать CRISPR-ассоциированная транспозаза CAST (CRISPR-associated transposase) цианобактерии Scytonema hofmanni. ShCAST катализирует РНК-управляемую транспозицию ДНК путем однонаправленной вставки ниже протоспейсера сегментов ДНК размером в 60-66 пар нуклеотидов .
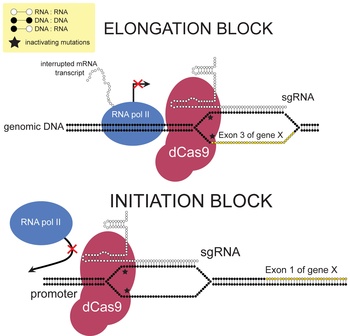
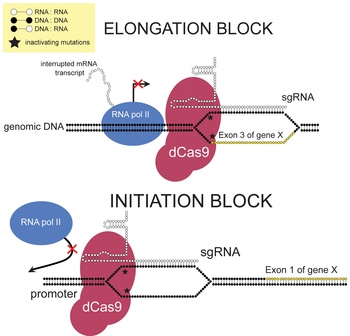
Использование dCas9
Соединив инактивированную молекулу dCas9, которая связывает ДНК, но не расщепляет её, с нуклеазой , удается получить нуклеазы и рестриктазы для высокоселективного разрезания ДНК . Разработан также способ избирательного эпигенетического перепрограммирования активности генов с помощью инактивированной молекулы dCas9, соединенной с ферментом, осуществляющим де метилирование ДНК . Причём такое перепрограммирование эпигенома можно проводить даже in vivo . Позднее выяснилось, что если укоротить управляющую РНК до 14-15 нуклеотидов, то молекула Cas9 теряет способность разрезать ДНК . Используя это свойство удалось создать систему для избирательной активации определённых генов in vivo и проверить её эффективность путем лечения мышей со смоделированными заболеваниями . У этого метода есть только одна проблема: обычно система CRISPR загружается в безвредный вирус, называемый аденоассоциированным вирусом (AAV) , который переносит систему в клетку. Но весь белок, состоящий из dCas9 и направляющей РНК, слишком велик, чтобы поместиться в один AAV. Чтобы обойти эту проблему, исследователи загрузили dCas9 в один вирус, а управляющую РНК — в другой . Cозданы трансгенные линии мышей для направленной регуляции генов in vivo путем редактирования эпигенома с помощью систем dCas9p300 и dCas9KRAB. Эти линии мышей являются удобными инструментами для манипулирования экспрессией генов in vivo с помощью различных направляющих РНК.
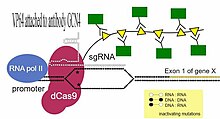
Повысить эффективность молекулы dCas9 позволяет повторяющийся полипептидный массив из повторяющихся антигенов , называемый SunTag, который может привлекать несколько копий антител , связанных с ферментом, осуществляющим эпигенетическое редактирование, или же с носителем флуоресцентной метки .
Еще одно применение dCas9 это программируемая модификация аминокислот в белках хроматина . Например искусственная гистон - протеинкиназа dCas9-dMSK1 позволяет осуществить гипер фосфорилирование серина 28 в (H3S28ph), играющего роль "пусковой кнопки" в избирательной активации промоторов , и таким образом, повысить экспрессию выбранных генов .
Новые способы доставки Cas9 в клетку
Основными требованиями к системе доставки Cas9 помимо высокой эффективности доставки является: (1) конструкция синтезирующая Cas9 не должна встраиваться в геном клетки и не должна быть в клетке постоянно чтобы не мешать работе клетки и не спровоцировать иммунных реакций; (2) средство доставки должно быть способно вместить достаточно большие по размерам фермент Cas9 или кодирующую его мРНК, а также одну или несколько направляющих РНК; (3) оно должно быть удобно для использования в виде инъекций; (4) такое средство вместе с Cas9 и направляющими РНК должно быть достаточно легко воспроизводимым для крупномасштабного производства лекарственного препарата для борьбы с распространенными болезнями. Таким критериям в отличие от вирусных систем доставки, отвечают липидные наночастицы. Так, например, была создана биодеградируемая система доставки Cas9 липидной наночастицей, которая позволила после однократного введения достичь in vivo более 97 % ингибирования уровня одного из белков сыворотки крови. При этом такое однократное введение, несмотря на временный характер системы доставки и компонентов системы редактирования, приводило к долговременному ингибированию продолжавшемуся в течение 12 месяцев . Для доставки используют также внеклеточные везикулы
Тем не менее продолжается разработка вирусных частиц для доставки Cas9 и . Одной из таких разработок является NanoMEDIC (nanomembrane-derived extracellular vesicles for the delivery of macromolecular cargo) NanoMEDIC эффективно индуцировал редактирование генома в различных типах клеток человека, таких как Т-клетки, моноциты, ИПСК, корковые нейроны, полученные из ИПСК, и миогенные клетки.
Поскольку существующие методы введения систем CRISPR-Cas в клетки с использованием вирусов-носителей и электрических импульсов, недостаточно эффективны для клеток, взятых непосредственно у пациентов (так называемых первичных клеток), предложено использовать комбинацию двух модифицированных пептидов, (одного из вируса ВИЧ и одного из вируса гриппа) CRISPR-Cas9 и Cas12a через внешнюю мембрану первичных клеток и в ядра, где находится большая часть клеточной ДНК. Это позволило сократить время инкубации и повысило эффективность редактирования генов до 98% . Метод назван PAGE (Peptide-Assisted Genome Editing).
Легче переносить в клетки для редактирования генома более компактные белки Cas, поскольку они могут быть упакованы в небольшие по объему средства доставки, такие как дезактивированный аденоассоциированный вирус (AAV). В качестве таких компактных белков можно использовать варианты Cas, обнаруженные в бактериофагах , например CRISPR-CasΦ, который вдвое меньше по молекулярной массе по сравнению с Cas9 или генноинженерный CasMINI, который несмотря на малые размеры, на клетках млекопитающих оказался столь же эффективен как и обычный Cas, и при этом лучше проникает в клетки
Модификация Cas9
Модификация Cas9 путем её слияния с хроматин-модулирующими пептидами, полученными из белков группы высокой подвижности и HMGB1 , гистона и хроматина , повышает её активность в несколько раз, особенно в отношении рефрактерных для неё участков хроматина. Эта стратегия слияния, называемая CRISPR-хром (англ. CRISPR-chrom), может быть использована для улучшения эффективности работы нуклеаз Cas9 при модификации генома .

CRISPR/Cas9 редактирование с праймером
- в этом методе используется нуклеаза Cas9 (модифицированная в никазу, так что она может создавать разрыв только в одной цепи ДНК) соединенная с обратной транскриптазой (RT) и вместо обычной направляющей РНК используется так называемая pegРНК (prime editing guid RNA - с праймером редактирующая гид РНК). Этот метод, по мнению авторов, является более точным и универсальным, чем все разработанные до сих пор альтернативы CRISPR .
Модульная платформа SSSavi на основе dCas9 для комбинаторного редактирования эпигенома
Cистема SSSavi представляет собой сменную и реконфигурируемую стыковочную платформу, объединенную с dCas9, что позволяет одновременно задействовать до четырех различных эффекторов, обеспечивая точный контроль состава эффекторов и их пространственного порядка. Пространственное предетерминированное упорядочение используемых эффекторов позволит более точно осуществлять регуляцию транскрипции.
Использование dCas9 для визуализации геномных последовательностей in situ
Исследователи разработали новый метод молекулярной визуализации с помощью РНК-направляемой эндонуклеазы CRISPR/dCas9 связанной с меткой. Технология позволила метить выбранные геномные последовательности в ядрах и хромосомах in situ. Метод назван RGEN-ISL. В отличие от классической флуоресцентной гибридизации in situ , RGEN-ISL не требует денатурации ДНК и, следовательно, обеспечивает лучшую сохранность структуры хроматина . Аналогичную функцию выполняет генетический инструмент, названный CRISPR-HOT (CRISPR–Cas9-mediated homology-independent organoid transgenesis), для цветной маркировки определенных генов в органоидах человека .
CARTRIDGE
CARTRIDGE (Cas-Responsive Translational Regulation Integratable into Diverse Gene control) технология использует белки Cas в качестве репрессоров и активаторов трансляции в клетках млекопитающих.
Эндонуклеазы, аналогичные Cas9
Cas12a
В отличие от Cas9 которая разрезает обе нити молекулы ДНК в одном и том же месте, оставляя тупые концы, оставляет одну нить длиннее другой, создавая липкие концы длиной 4-5 нуклеотидов , что повышает эффективность генетических вставок и их специфичность.
Fanzor
Fanzor это программируемая РНК-управляемая эндонуклеаза эукариот — организмов, включающих грибы, растения и животных. В отличие от белков CRISPR, ферменты эндонуклеазы Fanzor кодируются в эукариотическом геноме в составе мобильных элементов . .
См. также
- CRISPR
- Искусственные рестриктазы
- Домен белка
- Искусственные факторы транскрипции
- Генетическая инженерия
- Трансдифференцировка с помощью CRISPR-опосредованного активатора
Ссылки
- Heler R. , Samai P. , Modell J. W. , Weiner C. , Goldberg G. W. , Bikard D. , Marraffini L. A. (англ.) // Nature. — 2015. — Vol. 519, no. 7542 . — P. 199—202. — doi : . — .
- ↑ Jinek M. , Chylinski K. , Fonfara I. , Hauer M. , Doudna J. A. , Charpentier E. // Science. — 2012. — 28 июня ( т. 337 , № 6096 ). — С. 816—821 . — ISSN . — doi : .
- Carlaw, T. M., Zhang, L. H., & Ross, C. J. (2020). CRISPR/Cas9 Editing: Sparking Discussion on Safety in Light of the Need for New Therapeutics. Human Gene Therapy, 31(15-16), 794-807. doi :
- ↑ Guglielmi, G. (2018). от 15 июня 2021 на Wayback Machine . Nature, 558(7708), 17-19. doi :
- . Дата обращения: 12 октября 2020. 24 июня 2021 года.
- Wetsman N. (2022). от 2 марта 2022 на Wayback Machine . The Verge.
- Mali Prashant, Esvelt Kevin M, Church George M. Cas9 as a versatile tool for engineering biology // Nature Methods. — 2013. — Vol. 10. — P. 957-963. — ISSN . — doi : .
- Mali Prashant , Aach John , Stranges P Benjamin , Esvelt Kevin M , Moosburner Mark , Kosuri Sriram , Yang Luhan , Church George M. // Nature Biotechnology. — 2013. — 1 августа ( т. 31 , № 9 ). — С. 833—838 . — ISSN . — doi : .
- Gilbert Luke A. , Larson Matthew H. , Morsut Leonardo , Liu Zairan , Brar Gloria A. , Torres Sandra E. , Stern-Ginossar Noam , Brandman Onn , Whitehead Evan H. , Doudna Jennifer A. , Lim Wendell A. , Weissman Jonathan S. , Qi Lei S. // Cell. — 2013. — Июль ( т. 154 , № 2 ). — С. 442—451 . — ISSN . — doi : . — .
- Esvelt Kevin M , Smidler Andrea L , Catteruccia Flaminia , Church George M. // eLife. — 2014. — 17 июля ( т. 3 ). — ISSN . — doi : .
- Cyranoski David , Reardon Sara. // Nature. — 2015. — 22 апреля. — ISSN . — doi : .
- Pogson M. , Parola C. , Kelton W. J. , Heuberger P. , Reddy S. T. (англ.) // Nature communications. — 2016. — Vol. 7. — P. 12535. — doi : . — .
- Komor A. C. , Kim Y. B. , Packer M. S. , Zuris J. A. , Liu D. R. (англ.) // Nature. — 2016. — doi : . — .
- Gaudelli, N. M., Komor, A. C., Rees, H. A., Packer, M. S., Badran, A. H., Bryson, D. I., & Liu, D. R. (2017). Programmable base editing of A• T to G• C in genomic DNA without DNA cleavage. Nature, 551(7681), 464-471. doi : PMC PMID
- Xie, J., Huang, X., Wang, X., Gou, S., Liang, Y., Chen, F., ... & Jin, Q. (2020). ACBE, a new base editor for simultaneous C-to-T and A-to-G substitutions in mammalian systems. BMC biology, 18(1), 1-14. doi : PMC PMID
- Liu, Z., Chen, S., Shan, H., Jia, Y., Chen, M., Song, Y., ... & Li, Z. (2020). Precise base editing with CC context-specificity using engineered human APOBEC3G-nCas9 fusions. BMC biology, 18(1), 111 doi : PMC PMID
- Billon, P., Bryant, E. E., Joseph, S. A., Nambiar, T. S., Hayward, S. B., Rothstein, R., & Ciccia, A. (2017). CRISPR-mediated base editing enables efficient disruption of eukaryotic genes through induction of STOP codons. Molecular cell, 67(6), 1068-1079. doi : PMC PMID
- Kluesner, M. G., Lahr, W. S., Lonetree, C. L., Smeester, B. A., Claudio-Vázquez, P. N., Pitzen, S. P., ... & Webber, B. R. (2020). CRISPR-Cas9 cytidine and adenosine base editing of splice-sites mediates highly-efficient disruption of proteins in primary cells. bioRxiv. doi :
- Levy, J. M., Yeh, W. H., Pendse, N., Davis, J. R., Hennessey, E., Butcher, R., ... & Liu, D. R. (2020). Cytosine and adenine base editing of the brain, liver, retina, heart and skeletal muscle of mice via adeno-associated viruses. Nature Biomedical Engineering, 4(1), 97-110. doi : PMC PMID
- Roy K. R. , Smith J. D. , Vonesch S. C. , Lin G. , Tu C. S. , Lederer A. R. , Chu A. , Suresh S. , Nguyen M. , Horecka J. , Tripathi A. , Burnett W. T. , Morgan M. A. , Schulz J. , Orsley K. M. , Wei W. , Aiyar R. S. , Davis R. W. , Bankaitis V. A. , Haber J. E. , Salit M. L. , St Onge R. P. , Steinmetz L. M. (англ.) // Nature Biotechnology. — 2018. — July ( vol. 36 , no. 6 ). — P. 512—520 . — doi : . — .
- . Дата обращения: 9 мая 2018. 10 апреля 2019 года.
- Strecker J., Ladha A., Gardner Z., et al., (2019). RNA-guided DNA insertion with CRISPR-associated transposases. Science, eaax9181 doi :
- Tsai S. Q., Wyvekens N., Khayter C., Foden J. A., Thapar V., Reyon D., Goodwin M. J., Aryee M. J., Joung J. K. // Nature Biotechnology. — 2014. — Vol. 32, no. 6. — P. 569—576. — doi : . — .
- Guilinger J. P. , Thompson D. B. , Liu D. R. (англ.) // Nature biotechnology. — 2014. — Vol. 32, no. 6 . — P. 577—582. — doi : . — .
- Wyvekens N. , Topkar V. V. , Khayter C. , Joung J. K. , Tsai S. Q. (англ.) // Human gene therapy. — 2015. — Vol. 26, no. 7 . — P. 425—431. — doi : . — .
- Xu X. , Tao Y. , Gao X. , Zhang L. , Li X. , Zou W. , Ruan K. , Wang F. , Xu G. L. , Hu R. (англ.) // Cell discovery. — 2016. — Vol. 2. — P. 16009. — doi : . — .
- Morita S. , Noguchi H. , Horii T. , Nakabayashi K. , Kimura M. , Okamura K. , Sakai A. , Nakashima H. , Hata K. , Nakashima K. , Hatada I. (англ.) // Nature biotechnology. — 2016. — doi : . — .
- Xu, X.; Hulshoff, M.S.; Tan, X.; Zeisberg, M.; Zeisberg, E.M. CRISPR/Cas Derivatives as Novel Gene Modulating Tools: Possibilities and In Vivo Applications. Int. J. Mol. Sci. 2020, 21(9), 3038;
- Dahlman J. E. , Abudayyeh O. O. , Joung J. , Gootenberg J. S. , Zhang F. , Konermann S. (англ.) // Nature Biotechnology. — 2015. — November ( vol. 33 , no. 11 ). — P. 1159—1161 . — doi : . — .
- Kiani S. , Chavez A. , Tuttle M. , Hall R. N. , Chari R. , Ter-Ovanesyan D. , Qian J. , Pruitt B. W. , Beal J. , Vora S. , Buchthal J. , Kowal E. J. , Ebrahimkhani M. R. , Collins J. J. , Weiss R. , Church G. (англ.) // Nature Methods. — 2015. — November ( vol. 12 , no. 11 ). — P. 1051—1054 . — doi : . — .
- ↑ Liao H. K. , Hatanaka F. , Araoka T. , Reddy P. , Wu M. Z. , Sui Y. , Yamauchi T. , Sakurai M. , O'Keefe D. D. , Núñez-Delicado E. , Guillen P. , Campistol J. M. , Wu C. J. , Lu L. F. , Esteban C. R. , Izpisua Belmonte J. C. (англ.) // Cell. — 2017. — 14 December ( vol. 171 , no. 7 ). — P. 1495—1507 . — doi : . — .
- Gemberling, M., Siklenka, K., Rodriguez, E., Eisinger, K., Barrera, A., Liu, F., ... & Gersbach, C. (2021). Transgenic mice for in vivo epigenome editing with CRISPR-based systems. bioRxiv.
- ↑ Morita, S., Horii, T., Kimura, M., & Hatada, I. (2020). Synergistic Upregulation of Target Genes by TET1 and VP64 in the dCas9–SunTag Platform. International journal of molecular sciences, 21(5), 1574. doi : PMC PMID
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., & Vale, R. D. (2014). A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 159(3), 635-646. doi : PMC PMID
- Li, J., Mahata, B., Escobar, M. et al. (2021). Programmable human histone phosphorylation and gene activation using a CRISPR/Cas9-based chromatin kinase. Nat Commun 12, 896,
- от 12 февраля 2021 на Wayback Machine . ScienceDaily, 9 February 2021
- Wang, M., Zuris, J. A., Meng, F., Rees, H., Sun, S., Deng, P., ... & Xu, Q. (2016). Efficient delivery of genome-editing proteins using bioreducible lipid nanoparticles. Proceedings of the National Academy of Sciences, 113(11), 2868-2873.
- Qiu, M., Glass, Z., Chen, J., Haas, M., Jin, X., Zhao, X., ... & Xu, Q. (2021). Lipid nanoparticle-mediated codelivery of Cas9 mRNA and single-guide RNA achieves liver-specific in vivo genome editing of Angptl3. Proceedings of the National Academy of Sciences, 118(10). PMID doi :
- Finn J. D. , Smith A. R. , Patel M. C. , Shaw L. , Youniss M. R. , van Heteren J. , Dirstine T. , Ciullo C. , Lescarbeau R. , Seitzer J. , Shah R. R. , Shah A. , Ling D. , Growe J. , Pink M. , Rohde E. , Wood K. M. , Salomon W. E. , Harrington W. F. , Dombrowski C. , Strapps W. R. , Chang Y. , Morrissey D. V. (англ.) // Cell Reports. — 2018. — 27 February ( vol. 22 , no. 9 ). — P. 2227—2235 . — doi : . — .
- Horodecka, K., & Düchler, M. (2021). CRISPR/Cas9: Principle, Applications, and Delivery through Extracellular Vesicles. International Journal of Molecular Sciences, 22(11), 6072. PMID PMC doi :
- Gee, P., Lung, M. S., Okuzaki, Y., Sasakawa, N., Iguchi, T., Makita, Y., ... & Wang, X. H. (2020). Extracellular nanovesicles for packaging of CRISPR-Cas9 protein and sgRNA to induce therapeutic exon skipping. Nature Communications, 11(1), 1-18. PMC PMID doi :
- Zhang, Z., Baxter, A. E., Ren, D., Qin, K., Chen, Z., Collins, S. M., ... & Shi, J. (2023). Efficient engineering of human and mouse primary cells using peptide-assisted genome editing. Nature Biotechnology, 1-11. PMID doi :
- Pausch P., Al-Shayeb1 B., Bisom-Rapp E, et al. (2020). CRISPR-CasΦ from huge phages is a hypercompact genome editor. Science. 369(6501), 333-337 doi :
- Xiaoshu Xu, Augustine Chemparathy, Leiping Zeng, Hannah R. Kempton, Stephen Shang, Muneaki Nakamura, Lei S. Qi, (2021). Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing, Molecular Cell, .
- Ding X. , Seebeck T. , Feng Y. , Jiang Y. , Davis G. D. , Chen F. (англ.) // The CRISPR Journal. — 2019. — February ( vol. 2 ). — P. 51—63 . — doi : . — .
- ↑ Anzalone, A. V., Randolph, P. B., Davis, J. R., Sousa, A. A., Koblan, L. W., Levy, J. M., ... & Liu, D. R. (2019). Search-and-replace genome editing without double-strand breaks or donor DNA. Nature, 1-1. doi :
- ↑ Geurts, M. H., de Poel, E., Pleguezuelos-Manzano, C., Oka, R., Carrillo, L., Andersson-Rolf, A., ... & Clevers, H. (2021). Evaluating CRISPR-based prime editing for cancer modeling and CFTR repair in organoids. Life Science Alliance, 4(10). PMID doi :
- Ledford, H. (2019). Super-precise new CRISPR tool could tackle a plethora of genetic diseases. Nature, 574(7779), 464-465 doi :
- от 23 октября 2019 на Wayback Machine . The Scientist
- Swain, T., Pflueger, C., Freytag, S., Poppe, D., Pflueger, J., Nguyen, T., ... & Lister, R. (2024). A modular dCas9-based recruitment platform for combinatorial epigenome editing. Nucleic Acids Research, 52(1), 474–491 doi :
- Ishii T. , Schubert V. , Khosravi S. , Dreissig S. , Metje-Sprink J. , Sprink T. , Fuchs J. , Meister A. , Houben A. (англ.) // The New Phytologist. — 2019. — May ( vol. 222 , no. 3 ). — P. 1652—1661 . — doi : . — .
- Artegiani, B., Hendriks, D., Beumer, J. et al. (2020). . Nat Cell Biol
- Yang, Q., Oost, K.C. & Liberali, P. (2020). . Nat Cell Biol
- . Дата обращения: 3 марта 2020. 3 марта 2020 года.
- Kawasaki, S., Ono, H., Hirosawa, M. et al. (2023). Programmable mammalian translational modulators by CRISPR-associated proteins. Nat Commun 14, 2243
- Makoto Saito, Peiyu Xu, Guilhem Faure, Samantha Maguire, Soumya Kannan, Han Altae-Tran, Sam Vo, AnAn Desimone, Rhiannon K. Macrae, Feng Zhang. (англ.) // Nature. — 2023-06-28. — P. 1–3 . — ISSN . — doi : . 29 июня 2023 года.
- Сухинов, Даниил . Naked Science (29 июня 2023). Дата обращения: 10 июля 2023. 29 июня 2023 года.
Литература
- Джагаров Д. Э. (2014). . «Химия и жизнь -XXI век» № 7
- Джагаров Д. Э. (2014). . Academia.edu
- Дарья Спасская (2018). . N+1
- Монография (2020). . под ред. С.М. Закияна, С.П. Медведева, Е.В. Дементьевой, В.В. Власова. Монография состоит из 26 глав, в которых авторы детально описывают протоколы применения CRISPR‐опосредованных систем для модификации геномов различных организмов: от дрожжей до культивируемых клеток человека.
- Гоглева А. А., Артамонова И. И. (2014). CRISPR-системы: структура и гипотетические функции. Природа 6 (2014), 16-21;
- Гоглева А. А., Артамонова И. И. (2014). CRISPR-системы: механизм действия и применения. Природа 7 (2014), 3-9.
- Артамонова И.(2014). «биомолекула.ру»
- Руководство по пониманию и использованию CRISPR (на англ) Загрузите бесплатную электронную книгу здесь или здесь ?
- . Editors: M. Tofazzal IslamKutubuddin Ali Molla, Copyright: 2021 ISBN: 978-1-0716-1657-4
- Asmamaw, M., & Zawdie, B. (2021). Mechanism and Applications of CRISPR/Cas-9-Mediated Genome Editing. Biologics: Targets & Therapy, 15, 353. PMID PMC doi :
- Boti, M. A., Athanasopoulou, K., Adamopoulos, P. G., Sideris, D. C., & Scorilas, A. (2023). Recent Advances in Genome-Engineering Strategies. Genes, 14(1), 129;
- Nuñez, J. K., Chen, J., Pommier, G. C., Cogan, J. Z., Replogle, J. M., Adriaens, C., ... & Weissman, J. S. (2021). Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell, 184(9), 2503-2519. PMID PMC doi :
- How To Conduct Successful CRISPR Experiments eBook Загрузите бесплатную электронную книгу здесь
- Bravo, J.P.K., Liu, MS., Hibshman, G.N. et al. (2022). Structural basis for mismatch surveillance by CRISPR–Cas9 . Nature. doi : Новая версия фермента названная SuperFi-Cas9 в 4000 раз реже вносит ошибочные (не целевые) разрезы, но при этом работает так же быстро, как природный Cas9.
- Watters, K. E., Kirkpatrick, J., Palmer, M. J., & Koblentz, G. D. (2021). . Pathogens and Global Health, 1-13. PMID doi :
- Liu, H., Wang, L., & Luo, Y. Blossom of CRISPR technologies and applications in disease treatment (англ.) // Synth Syst Biotechnol : journal. — 2018. — Vol. 3 , no. 4 . — P. 217—228 . — doi : . — .
- Li, B., Niu, Y., Ji, W., & Dong, Y. Strategies for the CRISPR-Based Therapeutics (англ.) // Trends in Pharmacological Sciences : journal. — 2020. — Vol. 41 , no. 1 . — P. 55-65 . — doi : .
- Kazuto Yoshimi, Yayoi Kunihiro, Takehito Kaneko, Hitoshi Nagahora, Birger Voigt, Tomoji Mashimo. ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes (англ.) // Nature Communications : journal. — Nature Publishing Group , 2016. — Vol. 7 . — P. 10431 . — doi : . — .
- . The real power of the biological tool lies in exploring how genomes work. Nature 531, 156—159 (10 March 2016) doi : (на англ.) Обзор разных способов применения Cas9. Хорошие иллюстрации.
- Edited by Jennifer Doudna, University of California, Berkeley; Prashant Mali, University of California, San Diego
- Slaymaker, I. M., Gao, L., Zetsche, B., Scott, D. A., Yan, W. X., & Zhang, F. Rationally engineered Cas9 nucleases with improved specificity (англ.) // Science : journal. — 2016. — Vol. 351 , no. 6268 . — P. 84—88 . — doi : .
- Kleinstiver B. P., Pattanayak V., Prew M. S., , & J. Keith Joung et al. High-fidelity CRISPR–Cas9 nucleases with no detectable genome-wide off-target effects (англ.) // Nature : journal. — 2016. — doi : .
- Mandegar M. A., Huebsch N., Frolov E. B., , & Conklin B. R. et al. CRISPR Interference Efficiently Induces Specific and Reversible Gene Silencing in Human iPSCs (англ.) // : journal. — 2016. — doi : .
- Davis, K. M., Pattanayak, V., Thompson, D. B., Zuris, J. A., & Liu, D. R. Small molecule–triggered Cas9 protein with improved genome-editing specificity (англ.) // Nature chemical biology : journal. — 2015. — Vol. 11 , no. 5 . — P. 316—318 . — doi : .
- Zetsche, B., Volz, S. E., & Zhang, F. A split-Cas9 architecture for inducible genome editing and transcription modulation (англ.) // Nature biotechnology : journal. — 2015. — Vol. 33 , no. 2 . — P. 139—142 . — doi : .
- Zlotorynski, E. Genome engineering: Structure-guided improvement of Cas9 specificity (англ.) // Nature Reviews Molecular Cell Biology : journal. — 2016. — P. 3—3 .
- Chu, V. T., Weber, T., Wefers, B., Wurst, W., Sander, S., Rajewsky, K., & Kühn, R. (2015). . Nature biotechnology. doi :
- van Erp PB et al.(2015). . Curr Opin Virol.;12:85-90. PMID
- Cong, L., & Zhang, F. (2015). In Chromosomal Mutagenesis. Methods in Molecular Biology Vol. 1239, 2015, pp 197–217. Springer New York.
- Belhaj, K., Chaparro-Garcia, A., Kamoun, S., Patron, N. J., & Nekrasov, V. (2015). . Current opinion in biotechnology, 32, 76-84. doi :
- Kennedy E.M., Cullen B.R. (2015). Virology, 479—480, 213—220 doi :
- Junwei Shi, Eric Wang, Joseph P Milazzo, Zihua Wang, Justin B Kinney, Christopher R Vakoc.(2015). . Nature Biotechnology,; doi :
- Michael Boettcher, Michael T. McManus (2015). . Molecular Cell, 58(4), p575-585 DOI
- Heidi Ledford. A powerful gene-editing technology is the biggest game changer to hit biology since PCR. But with its huge potential come pressing concerns. (англ.) // NATURE : journal. — 2015. — Vol. 522 . — P. 20—24 . — doi : .
- см. также: Chari R, Mali P, Moosburner M, Church GM (2015). Unraveling CRISPR-Cas9 genome engineering parameters via a library-on-library approach. Nature Methods (in press).
- Amanda Andersson-Rolf , & Bon-Kyoung Koo et al. One-step generation of conditional and reversible gene knockouts. (англ.) // Nature Methods : journal. — 2017. — doi : . CRISPR-FLIP, a strategy that provides an efficient, rapid and scalable method for biallelic conditional gene knockouts in diploid or aneuploid cells.
- SØREN HOUGH (2017). .
- Nakajima, K., Zhou, Y., Tomita, A., Hirade, Y., Gurumurthy, C. B., & Nakada, S. (2018). . Genome research, 28(2), 223—230. PMC doi :
- Qiu, X. Y., Zhu, L. Y., Zhu, C. S., Ma, J. X., Hou, T., Wu, X. M., … & Zhu, L. (2018). . ACS synthetic biology, 7(3), 807—813. doi : PMID
- Ting Wang, Yong Liu, Huan-Huan Sun, Bin-Cheng Yin, Bang-Ce Ye.(2019). An RNA-Guided Cas9 Nickase-Based Method for Universal Isothermal DNA Amplification. Angewandte Chemie International Edition, doi :
- Smith, C. J., Castanon, O., Said, K., Volf, V., Khoshakhlagh, P., Hornick, A., ... & Myllykallio, H. (2019). . bioRxiv 574020 doi : Метод позволяет одновременно редактировать более 10 000 локусов в клетках человека.
- Hirosawa, M., Fujita, Y., & Saito, H. (2019). . ACS synthetic biology. 8(7), 1575-1582 PMID
- Wang, D., Zhang, F., & Gao, G. (2020). CRISPR-Based Therapeutic Genome Editing: Strategies and In Vivo Delivery by AAV Vectors. Cell, 181(1), 136-150. PMID doi :
- Alagoz, M., & Kherad, N. (2020). : CRISPR therapy. International Journal of Molecular Medicine.
- STEVEN LEVY (2020). . WIRED
- Timothy R. Abbott, Girija Dhamdhere, Yanxia Liu et al., (2020). Development of CRISPR as an Antiviral Strategy to Combat SARS-CoV-2 and Influenza. Cell
- Xie H, Ge X, Yang F, Wang B, Li S, Duan J, et al. (2020). High-fidelity SaCas9 identified by directional screening in human cells. PLoS Biol 18(7): e3000747.
- Armando Casas-Mollano J., Zinselmeier M. H. , Erickson S.E. , and Smanski M.J. (2020). . CRISPR J.; 3(5): 350–364 doi : PMC
- Horodecka K, Düchler M. CRISPR/Cas9: Principle, Applications, and Delivery through Extracellular Vesicles. International Journal of Molecular Sciences. 2021; 22(11):6072.
- Denes CE, Cole AJ, Aksoy YA, Li G, Neely GG, Hesselson D. (2021). Approaches to Enhance Precise CRISPR/Cas9-Mediated Genome Editing. International Journal of Molecular Sciences. 22(16):8571.
- Pan, C., Wu, X., Markel, K. et al. (2021). CRISPR–Act3.0 for highly efficient multiplexed gene activation in plants. Nat. Plants
- Park, J., Yoon, J., Kwon, D., Han, M. J., Choi, S., Park, S., ... & Choe, S. (2021). Enhanced genome editing efficiency of CRISPR PLUS: Cas9 chimeric fusion proteins. Scientific Reports, 11(1), 1-9. PMID PMC doi :
- Corsi, G. I., Gadekar, V. P., Gorodkin, J., & Seemann, S. E. (2021). CRISPRroots: on-and off-target assessment of RNA-seq data in CRISPR–Cas9 edited cells. Nucleic Acids Research, 50(4), e20 PMID doi :
- Nambiar, T. S., Baudrier, L., Billon, P., & Ciccia, A. (2022). CRISPR-based genome editing through the lens of DNA repair. Molecular Cell, 82(2), 348-388. PMID PMC (available on 2023-01-20) doi :
- Amendola, M., Brusson, M., & Miccio, A. CRISPRthripsis: The Risk of CRISPR/Cas9-induced Chromothripsis in Gene Therapy. Stem Cells Translational Medicine, 11(10), 1003–1009 PMID PMC doi :
- Carusillo, A., Haider, S., Schäfer, R., Rhiel, M., Türk, D., Chmielewski, K. O., ... & Mussolino, C. (2023). A novel Cas9 fusion protein promotes targeted genome editing with reduced mutational burden in primary human cells. Nucleic Acids Research, 51(9), 4660-4673. PMID PMC doi :
- Passos, G. A. (2023). . Springer Nature. 209 стр. Книга фокусируется на применении системы Crispr для редактирования генома клеток человека (in vitro и in vivo) и модельных организмов, используемых в биомедицинских исследованиях.
- Tyumentseva, M., Tyumentsev, A., & Akimkin, V. (2023). CRISPR/Cas9 Landscape: Current State and Future Perspectives. International Journal of Molecular Sciences, 24(22), 16077. doi :
Ссылки
- 1CRISPR/Cas9 Genome Editing : Application Guide : [ англ. ] : [ 5 сентября 2015 ]. — OriGene. — 53 p.
- 2020-03-21
- 1