Искусственный геном
- 1 year ago
- 0
- 0

Прое́кт «Ра́ковый гено́м» ( англ. Cancer Genome Project ) — исследовательский проект при Институте Сенгера , нацеленный на поиск мутаций , ведущих к развитию раковых заболеваний человека. Проект был запущен в 2000 году, и возглавляется им и его коллегой Питером Кэмпбеллем . «Раковый геном», как и аналогичный проект « Атлас Ракового Генома » ( англ. The Cancer Genome Atlas (TCGA) ) при Национальном институте рака в США , ставит своей целью усовершенствование диагностики, лечения и предотвращения опухолевых заболеваний посредством изучения молекулярных основ их развития .
С помощью методов высокопроизводительного секвенирования исследуется генетический материал из опухолей различных типов. Результаты публикуются в базе данных Catalogue of Somatic Mutations in Cancer (COSMIC) , самой полной из имеющихся баз данных о генетических изменениях в раковых клетках . По состоянию на 4 мая 2019 года, выпущена 88-я версия от 19 марта 2019 года . База обновляется раз в три месяца .
Совместно с Центром молекулярной терапии при Массачусетской больнице общего профиля в Бостоне, «Раковый геном» развивает базу данных Genomics of Drug Sensitivity in Cancer (GDSC) , содержащую информацию о скрининге противоопухолевых препаратов. Целью коллаборации является усовершенствование лечения раковых заболеваний на основе предсказания возможной реакции организма пациента на противоопухолевый препарат .
Исследовательская группа, работающая над проектом «Раковый геном», публикует на портале Института Сенгера разработанные ими программы для работы с данными о раковых геномах: BioView и AutoCSA (программы для детекции мутаций), Allele-Specific Copy number Analysis of Tumors (ASCAT) и другие . На странице проекта можно найти публикации исследовательской группы, большинство из которых находится в открытом доступе .
Catalogue of Somatic Mutations in Cancer (COSMIC) — одна из самых больших и полных онлайн баз данных соматических мутаций , свойственных различным типам опухолевых заболеваний человека. Обновляется раз в три месяца. По состоянию на 4 мая 2019 года, выпущена 88-я версия от 19 марта 2019 года .
Система пополняется двумя способами. Первый — ручной сбор данных курирующими экспертами; источником служат рецензируемые издания, из которых эксперты отбирают информацию и вносят её в систему. Второй — полуавтоматический сбор данных: загружаются данные по масштабному скринингу больших раковых геномов и экзомов из баз The Cancer Genome Atlas ( ) и International Cancer Genome Consortium (ICGC) .
Накопленная COSMIC информация о 2500 раковых заболеваниях человека делает возможными выводы о соответствии мутаций определённому типу опухоли. Представленные в COSMIC данные подтверждаются научными публикациями (около 20 000 статей PubMed ), тщательно отбираемыми кураторами (около 30 % публикаций отвергаются) . Все данные, представленные в COSMIC, доступны для скачивания в нескольких форматах после регистрации на сайте .
База данных была запущена 4 февраля 2004 года и содержала информацию о четырёх генах: HRAS, KRAS , NRAS и BRAF . К концу 2005 года в базе данных были представлены последовательности 529 генов из 115 327 опухолей, содержащие 20 981 мутацию . К концу августа 2009 года COSMIC включал данные, полученные в ходе 1,5 миллионов экспериментов и описывающие более 90 000 мутаций в 13 423 генах почти в 370 000 типов опухолей . 48-я версия COSMIC, выпущенная в июле 2010, содержала 141 212 мутаций, определённых в ходе более чем 2,76 миллионов экспериментов для более 50 000 типов опухолей . 70-я версия (август 2014 года), включала уже 2 002 811 точечных мутаций в кодирующих последовательностях, определённых для свыше миллиона опухолей человека. Кроме мутаций данного типа, в 70-й версии уже описаны более 6 миллионов мутаций в некодирующих областях , 10 534 слияний генов, 61 299 геномных перестроек, 695 504 участков с нарушенной копийностью и более 60 миллионов случаев изменения экспрессии генов .

COSMIC предоставляет доступ ко всем данным в системе. Система включает ряд инструментов: Cancer Browser, Genome Browser, GA4GH Beacon, CONAN .
Cancer Browser позволяет проанализировать информацию о свыше 2500 типах раковых заболеваний. Выбрав тип ткани (44 вариантов в версии 80) и её гистологии, можно получить данные о встречаемости в них мутаций. К примеру, можно получить график, отображающий наиболее часто мутирующие гены, аннотированные в Cancer Gene Census , и частоты их мутации, определяемые как отношение числа образцов с мутациями (показано синим) к числу проанализированных образцов (показано красным) .
Genome Browser позволяет осуществить поиск информации о мутациях в интересующем гене. Так, для гена транскрипционного фактора p53 (TP53) в 72-й версии COSMIC представлены данные, основанные на анализе 104 011 индивидуальных образцов и 27 507 из них несли мутации данного гена. Также приведены 2213 статьи PubMed с описанием и ссылками на источники .
GA4GH (Глобальный альянс геномики и здоровья, англ. Global Alliance for Genomics and & Health ) Beacon — это сервис для предоставления COSMIC генетических данных сообществом .
CONAN — инструмент для анализа вариаций числа копий генов .
Cancer Gene Census содержит список генов (723 на май 2019, версия 88) , их отношение к возникновению заболевания, типы мутаций, приводящие к дисфункции гена в раковых клетках, а также типы опухолей, в которых наблюдаются данные мутации .
Отбор кандидатов начинается с поиска паттернов соматических мутаций, приводящих к развитию рака. Для выявления функции выбранного гена и его влияния на развитие рака далее проводится тщательный обзор литературы. На этой стадии ген классифицируется как онкоген , ген-супрессор опухоли , либо как обладающий обеими функциями. Если функция гена проявляется в результате слияния с другим геном, он обозначается как ( англ. fusion gene ) .
В зависимости от того, насколько хорошо доказано участие гена в онкогенезе, гены классифицируются на два «яруса» ( англ. tiers ). Гены Яруса 1 (Tire 1) характеризуются паттернами мутаций, участие и функции которых в этиологии опухоли считаются прочно доказанными. Для отнесения гена к Ярусу 1 необходимо наличие как минимум двух публикаций от двух независимых групп, которые описывают соматическую мутацию гена в как минимум одном типе рака. В Ярус 2 (Tire 2) попадают гены, участие которых в развитии рака доказано на обширных литературных данных, однако информации, подтверждающей последствия мутации существует недостаточно .
Последняя версия CGC содержит 723 гена (576 Яруса 1, 147 Яруса 2). Из них 562 определяются как онкогены и/или онкосупрессоры, 132 гена с неизвестной функцией проявляют свою активность в результате слияния, 30 генов не были отнесены ни к одной из групп .
В 86-м обновлении была добавлена возможность визуализации данных об особенностях участия гена в развитии опухоли. В краткой записи приводится описание функции самого гена и его связь с десятью основными признаками рака .
COSMIC-3D — интерфейс для изучения раковых мутаций по трехмерной структуре белка; впервые представлен в 80 выпуске COSMIC , подготовлен в партнерстве с with Astex Pharmaceuticals (Cambridge, UK). Инструмент показывает трехмерную визуализацию более чем 8000 белков, на которых отмечены мутации из базы COSMIC, а также их частота и эффект .
Вначале работы программы мутация картируется на последовательность белка из UniProt, затем на PDB-структуру белка при помощи SIFTS UniProt-to-PDB. Кроме того, COSMIC-3D может искать пересечения между местом мутации, приводящей к развитию рака, известными сайтами связывания низкомолекулярных веществ и сайтами связывания лекарств, предсказанных fPocket. Полученные данные могут позволить производить молекулы, специфически связывающиеся с мутированными белками .
COSMIC Cell Line Project содержит информацию о полном секвенировании экзомов более чем 1015 различных раковых клеточных линий . Экзомы добавляются непосредственно после их получения, до публикации . Использование COSMIC Cell Line Project позволяет осуществлять более осмысленный выбор клеточных линий для исследований и более качественную интерпретацию результатов .

Genomics of Drug Sensitivity in Cancer (GDSC) — дополнительный ресурс проекта «Раковый геном», содержащий информацию о чувствительности свыше 700 опухолевых клеточных линий к более чем 140 противораковым препаратам, а также предоставляющий данные о корреляции между мутациями и чувствительностью к препаратам . Представленные в GDSC данные были получены высокопроизводительным скринингом, проводимым в рамках проекта «Раковый геном» Институтом Сенгера и Центром молекулярной терапии при Массачусетской больнице общего профиля в Бостоне для коллекции из свыше 1000 клеточных линий. Соединения, выбранные для скрининга, включают как препараты одобренные для применения в клинике и проходящие клинические испытания, так и препараты ещё находящиеся в разработке. Данные соединения воздействуют на целый ряд мишеней, в том числе на компоненты сигнальных путей с участием тирозин-киназных рецепторов, контроля клеточного цикла и системы ответа на повреждение ДНК .
Неотъемлемым свойством GDSC является интеграция информации как о раковых геномах, так и о чувствительности опухолевых клеток к препаратам. С целью выявления маркеров для предсказания ответа на препарат применяются 2 дополняющих друг друга аналитических подхода .
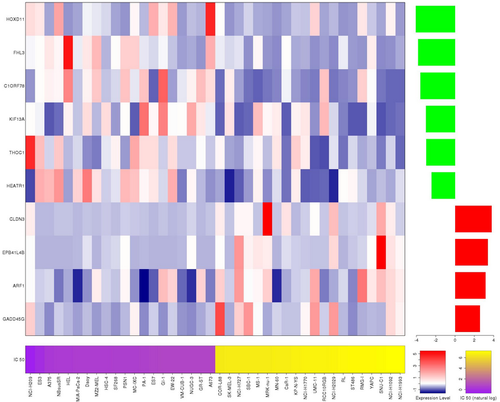
Первый поход — multivariate analysis of variance (MANOVA), разновидность метода ANOVA , используется для определения корреляции между чувствительностью к препарату (по IC50 и наклону кривой доза-эффект ) и изменениями в геноме клетки ( точечными мутациями , амплификациями или делециями генов и др.). При этом для каждой пары препарат-ген MANOVA определяется характер эффекта и статистическая значимость связи. Данные представляются в виде . Размер отображаемых кружков соответствует числу событий, взятых для анализа. Наведение на кружок позволяет узнать информацию, касающуюся размера выборки (число клеточных линий), эффект (во сколько раз чувствительность к препарату усиливается или уменьшается) и p-value .
Второй подход — применение штрафной функции «elastic net». Данные, анализируемые с помощью данной штрафной функции, помимо подаваемых MANOVA, включают полногеномные транскрипционные профили и типы тканей. Функция «elastic net» определяет характеристики, связанные с определённым ответом на препарат (значением IC50 ) той или иной клеточной линии. Данные представляются в виде «elastic net plot» — тепловой карты и гистограммы. Тепловая карта отображает мутации 20 наиболее устойчивых и 20 наиболее чувствительных к данному препарату клеточных линий. Цвета карты отображают экспрессию и копийность (синий — низкая, красный — высокая). Гистограмма показывает изменение чувствительности к препарату, соответствующее мутации (увеличение — красный, снижение — зелёный) .
Исследования, проводимые участниками проекта «Раковый геном», пополняют знания, накопленные о модификациях генома, ведущих к опухолевым трансформациям , позволяя усовершенствование методов предсказания, диагностики и терапии. Работы посвящены исследованию самых различных типов опухолей. Кроме того, ведётся поиск маркеров раковых заболеваний и разработка систем для проведения исследования на модельных организмах .
Одним из направлений работы проекта «Раковый геном» является исследование рака молочной железы . Так, анализ соматических мутаций 21-го типа рака молочной железы выявил наличие регионов с повышенной частотой мутаций (такой регион был назван «kataegis»). Данные области колокализировались с соматическими перестройками, и замены в данных регионах практически всегда приходились на цитозин TpC динуклеотидов. За основу данного явления была предложена работа представителей APOBEC семейства цитидин деаминаз . Дальнейшие исследования послужили в пользу данной гипотезы, показав, что типы рака молочной железы с делецией APOBEC3B характеризуются большим числом мутаций в регионах «kataegis» .
Анализ точечных мутаций и вариаций числа копий генов для 100 разновидностей рака молочной железы выявил многочисленные нарушения в геноме, в частности 9 новых генов (AKT2, ARID1B, CASP8, CDKN1B , MAP3K1, MAP3K13, NCOR1, SMARCD1 и TBX3) были ассоциированы с данным типом рака . При этом мутации в генах MAP3K1, MAP2K4, MAP3K13 и AKT2 нарушают активацию сигнального пути JUN, снижение активности которого показано для более чем 50 % типов рака молочной железы . Для мутаций в ряде генов (ARID1B, CASP8, MAP3K1, MAP3K13, NCOR1, SMARCD1 и CDKN1B ) было выявлено усиление экспрессии укороченных изоформ белков, что предполагает, что исходные формы данных белков могут относиться к супрессорам развития трансформаций. Кроме того, была показана связь между пониженной экспрессией рецепторов эстрогена и ускоренным накоплением мутаций с возрастом .
Скрининг около 3500 генов выявил несколько новых генов, мутации которых ведут к развитию гипернефроидной опухоли почки, редкой разновидности рака почки . Данные гены включают кодирующие деметилазы UTX (KDM6A) и JARID1C (KDM5C) и кодирующий метилазу ген SETD2 . Данные ферменты модифицируют ключевые остатки лизина гистона H3 , влияя на структуру хроматина и транскрипцию генов. При этом совместно данные мутации присутствует менее чем в 15 % случаев развития гипернефроидной опухоли почки, предполагая существование ещё не выявленных генов. Более поздние эксперименты по секвенированию экзома выявили ген PBRM1, компонент комплекса SWI/SNF, отвечающего за перестройки хроматина, в качестве одного из самых важных генов, мутации которого в 41 % случаев приводят к гипернефроидной опухоли почки .
Результаты секвенирования, проведенного для клеточной линии NCI-H209 злокачественной мелкоклеточной опухоли легкого , выявили 22 910 соматических замен, в том числе 132 в кодирующих участках, ассоциированных с табакокурением . При этом для клеточной линии NCI-H209 была показана дупликация 3-8 экзонов CHD7, а для двух других линий мелкоклеточной опухоли легкого продемонстрировано слияние генов PVT1 и CHD7, в совокупности предполагая, что мутации гена CHD7 способствуют развитию заболевания .
Было показано, что раку поджелудочной железы свойственны разнообразные перестройки, приводящие к дисфункции теломер и нарушению контроля клеточного цикла , в частности к поломкам перехода из фазы G1 в S-фазу. Это запускает амплификацию онкогенов , что преимущественно происходит на ранних стадиях развития заболевания .
Важным направлением работы исследовательской группы является изучение одного из наиболее распространенных видов рака — рака толстой кишки (колоректальной карциномы) .
Число комбинаций различных генетических изменений не позволяет выявить функциональный вклад каждого потенциального гена в развитие опухоли. Поэтому, несмотря на то что определение геномных изменений в индивидуальных опухолях возможно с высокой точностью и по относительно низкой цене, эти данные сложно интерпретировать с точки зрения прогнозирования развития заболевания и поиска нужных лекарств, для этого требуется наличие модельной системы для анализа генотип-фенотипической корреляции. Такими модельными системами могут служить трехмерные органоиды . В данном исследовании с использованием Lgr5 стволовых клеток (располагаются в криптах) были получены опухолевые органоидные культуры 20 пациентов с колоректальной карциномой. Было показано, что спектр генетических изменений, а также анализ генной экспрессии в органоиде согласуется с таковыми в самой опухоли. Кроме того, органоид поддается лекарственному скринингу. В качестве примера было приведено влияние ингибитора поркупина на мутантов по гену RNF43, в результате чего рост мутанта прекращался .
Для выяснения природы внутриопухолевого разнообразия были охарактеризованны органоиды, полученные из множества единичных клеток из трёх различных опухолей колоректальной карциномы, а также из стволовых клеток крипт. Было показано, что во всех раковых клетках число соматических мутаций в несколько раз больше, чем в нормальных клетках, причем большинство мутаций оказались приобретены в ходе финальной клональной экспансии рака, посредством процессов, отсутствующих в нормальных клетках. Также наблюдались различия в транскриптомах и метиломах внутриопухолевых клеток. Данные различия проявляются в разной реакции даже близкородственных клеток на противораковые препараты .
Анализ данных, представленных в GDSC, выявил ряд потенциальных биомаркеров для терапевтического использования. В частности, было показано, что мутации TP53 коррелируют с устойчивостью к нутлину-3a, ингибитору MDM2 убиквитинлигазы p53 . Амплификация CCND1 (CyclinD1) или потеря SMAD4 были ассоциированы с повышенной чувствительностью к многочисленным ингибиторам семейства EGFR , например к лапатинибу . Потеря SMAD4 сопровождалась повышенной экспрессией EGFR. Инактивация STK11, репрессора mTOR , коррерировала с повышением чувствительности к ингибитору белка теплового шока HSP90, 17-AAG. Кроме того, посредством выявления транслокации EWS-FLI1 в качестве маркера чувствительности к ингибиторам PARP , была отмечена повышенная чувствительность к соединениям данного характера клеток саркомы Юинга , что предлагает новые методы борьбы с данным заболеванием .
Круглый червь Caenorhabditis elegans , широко используемый модельный организм , был предложен в роли экспериментальной системы для изучения влияния канцерогенов и дефектов системы репарации ДНК . Проведено секвенирование полных геномов 183 популяций C. elegans с 17 разновидностями генетических бэкграундов ( дикий тип и особи с нокаутированными генами репарации ДНК и системы ответа на повреждения ДНК), в результате чего были выявлены 1559 замен, 406 вставок и 281 геномная перестройка. Полученные результаты были сопоставлены с данными, накопленными о канцерогенезе человека .
Проанализировано действие таких канцерогенов , как афлатоксин B1, хлорметин и цисплатин на C. elegans . Интересно, что мутагенные свойства цисплатина наиболее проявлялись в мутантах xpf-1, предполагая важную роль данного гена в защите клеток от данного ДНК-повреждающего агента. В целом же результаты экспериментов соответствовали ранее известным данным о действии рассматриваемых канцерогенов , продемонстрировав возможность применения экспериментальной системы для изучения менее исследованных веществ. В частности, более 240 соединений рассматриваются в качестве потенциальных канцерогенов . Однако накопленной информации об их действии недостаточно, и использование экспериментальной системы представляет собой удобный способ для её получения .
Анализ геномов клеток эпителия пищевода показал, что некоторые мутации приводят к обогащению эпителия клонами мутировавших клеток. Так, мутанты по гену NOTCH1, который отвечает за взаимодействие контактирующих клеток, могут составлять до 80 % клеток эпителия в людях среднего и старшего возраста, а мутанты по гену TP53 — до 37 %. Подобные результаты были получены всего для 14 мутаций, участвующих в раковом перерождении. При этом частота мутантов по гену NOTCH1 у здоровых людей была больше, чем у больных раком пищевода. Это позволяет предположить, что некоторые мутации могут приводить к бурному клональному размножению, но уменьшать шанс ракового перерождения .
Результаты секвенирования 112 первичных и метастатических опухолей простаты привели к обнаружению 22 новых генов, приводящих к развитию рака. Кроме того, было обнаружено ещё 2 не кодирующих белки участка, ассоциированных с раком простаты. Также было выяснено, в каком порядке эти мутации происходят в развивающихся опухолях. Анализ открытых и уже известных мутаций показал, что 11 из них являются мишенями имеющихся лекарств, 7 — мишенями лекарств, проходящих клинические испытания, для 13 проводятся исследования и разработка лекарств и 49 потенциально могут быть мишенями будущих препаратов .
Для того, чтобы точнее оценивать искажения, вызванные использованием системы CRISPR-Cas для анализа генов путем их нокаутирования, был создан новый алгоритм CRISPRcleanR. Было учтено систематическое недооценивание logFC для участков, содержащих много копий мишеней направляющей РНК, а также некоторые другие отклонения. Новый алгоритм уменьшает частоту ложноположительных находок, оставляя частоту истинных положительных находок на прежнем уровне . CRISPRcleanR находится в свободном доступе и представлен как пакет R и пакет Python .