Interested Article - GENCODE
- 2020-07-07
- 1
GENCODE — проект геномных исследований, являющийся составной частью проекта « Энциклопедия элементов ДНК » (ENCODE) .
GENCODE был создан в рамках пилотной фазы проекта ENCODE с целью определить и картировать все белок -кодирующие гены из библиотеки ENCODE . Сейчас проект нацелен на создание энциклопедии генов и их вариантов с полным описанием их структурных характеристик в геномах человека и мыши с помощью методов вычислительного анализа, ручной аннотации и экспериментальной проверки .
Конечная цель проекта — создание базы аннотаций, включающей все белок-кодирующие локусы с альтернативными транскриптами , некодирующие локусы с обнаруженными транскриптами и псевдогены .
История
В сентябре 2003 года Национальный институт исследований генома человека ( ) открыл публичный исследовательский консорциум ENCODE для реализации проекта по определению всех функциональных элементов человеческого генома. Этот проект является продолжением проекта «Геном человека» ( англ. The Human Genome Project ) , запущенного в 1990 году Национальной организацией здравоохранения США ( NIH ). Целью проекта была расшифровка последовательности ДНК человека. В 2003 году были опубликованы результаты, и научное сообщество высказало заинтересованность в исследовании функциональных элементов генома человека для лучшего понимания механизмов развития некоторых заболеваний. Для этого и был запущен проект ENCODE. Он был разделен на три фазы: пилотная (начальная) фаза, разработка методологии и продуктивная фаза . В ходе пилотной фазы планировалось исследовать около 30 Mb генома человека , а полученные результаты учесть в дальнейшем при анализе остального человеческого генома . Для картирования на этот фрагмент генома известных белок-кодирующих генов был создан проект GENCODE .
В апреле 2005 года была выпущена первая версия GENCODE с аннотацией 44 локусов человеческого генома . В ней было описано 416 известных геномных локусов, 26 новых белок-кодирующих локусов, 82 транскрибирующихся и 170 псевдогенных локусов . Во втором релизе (14 октября 2005 года) была обновлена и подтверждена информация об аннотированных раньше локусах, в основном, благодаря экспериментальным данным и RT-PCR .
В июне 2007 года пилотная фаза ENCODE была завершена . Проект был признан удачным, и институт Сэнгера (Welcome Trust Sanger Institute) получил грант от NHGRI для масштабирования проекта GENCODE .
В 2012 году был выпущен крупнейший релиз GENCODE 7 (на основе данных на момент декабря 2011 года), в котором была скомбинирована автоматическая аннотация Ensembl и аннотация вручную. С 2013 года GENCODE был удостоен второго гранта на продолжение работы по аннотации генома человека, а также на аннотацию мышиного генома для сравнительных исследований геномов мыши и человека .
В апреле 2018 года была выпущена версия GENCODE 28 (содержащая данные, обработанные до ноября 2017 года) .
Задачи
Задачи, решавшиеся в проекте GENCODE, вставали перед научным сообществом по мере расширения познаний в области генетики. Как правило, эти задачи были связаны с уточнением определений генетических терминов и с изучением функций геномных участков, ранее не подвергавшихся близкому рассмотрению. Ниже приведены примеры интересных вопросов и тем, которые исследуются в рамках GENCODE .
Определение понятия «ген»
Задача определения понятия « ген » стоит перед учеными на протяжении всего времени с тех пор, как исследователи задумались о вопросах наследственности. В 1900-х годах ген рассматривался как некая дискретная единица наследственности, затем ген стали считать основой для биосинтеза белка , а в последнее время это понятие расширили до геномного фрагмента, транскрибирующегося в РНК . Хотя определение гена претерпело значительные изменения за последний век, оно осталось сложным и противоречивым предметом обсуждения для многих ученых. В ходе развития проектов ENCODE и, в частности, GENCODE, были подробнее описаны ещё более проблематичные аспекты определения — такие как альтернативный сплайсинг , межгенные участки, а также сложные паттерны рассеянной регуляции , консервативность некодирующих участков и обилие генов, продуцирующих некодирующие РНК . Поскольку глобальной целью проекта GENCODE является создание энциклопедии генов и генных вариантов, эти проблемы поставили проект перед необходимостью дать обновленное определение понятия гена .
Псевдогены
Псевдогены — это белок-кодирующие (или сходные с ними) последовательности ДНК, в которых произошла делеция или сдвиг рамки считывания . В большинстве геномных баз данных их упоминают как побочные продукты аннотации более привычных белок-кодирующих последовательностей. Однако недавний анализ показал, что некоторые из псевдогенов не просто экспрессируются, но и функционируют, играя роль в различных биологических процессах . Чтобы разобраться со всеми сложностями описания псевдогенов, в рамках GENCODE исследователи создали онтологию псевдогенов с использованием автоматических, ручных и экспериментальных методов, чтобы связать воедино их различные свойства, в том числе свойства последовательности, эволюцию и возможную биологическую функцию . Количество аннотированных псевдогенов растет с каждой новой версией GENCODE (см. ).
Длинные некодирующие РНК (lncRNA)
Одной из ключевых областей исследования проекта GENCODE является изучение биологического значения длинных некодирующих РНК (lncRNA). Для более глубокого понимания и изучения экспрессии lncRNA у человека, в рамках GENCODE был запущен подпроект для разработки пользовательских микрочипов для количественного определения таких транскриптов в аннотации lncRNA . Некоторое количество подобных платформ было создано с помощью системы Agilent Technologies eArray, они доступны в стандартном формате .
Датасет длинных некодирующих РНК, представленный в GENCODE (в частности, в версии GENCODE 7), считается самым большим из всех представленных датасетов lncRNA. При этом он мало перекрывается с другими существующими датасетами . Транскрипты, аннотированные как lncRNA, далее могут быть классифицированы на следующие типы на основе их положения в геноме относительно белок-кодирующих генов:
- Антисмысловая РНК : локус, для которого был найден хотя бы один транскрипт, перекрывающийся с экзоном белок-кодирующего гена на противоположной цепи, или есть опубликованные данные об антисмысловой регуляции какого-либо гена;
- Длинная межгенная некодирующая РНК ( );
- «Перекрывающийся»: локус, содержащий белок-кодирующий ген внутри интрона на той же цепи;
- «Интронный»: локус, расположенный внутри интрона, но не перекрывающийся с экзонами на той же цепи;
- Процессированный транскрипт: локус, для которого ни один транскрипт не содержит открытую рамку считывания , и который не может быть отнесен ни к одной из предыдущих категорий из-за сложной структуры.
Основные участники
В таблице приведены институты, чье участие было анонсировано на сайте .
| Пилотная фаза | Масштабирование проекта | Вторая фаза (текущая) |
|---|---|---|
| Институт Сэнгера , Кэмбридж, Великобритания | Институт Сэнгера , Кэмбридж, Великобритания | Институт Сэнгера , Кэмбридж, Великобритания |
| Муниципальный Институт Медицинских Исследований (IMIM), Барселона, Каталония | , Барселона, Каталония | , Барселона, Каталония |
| Университет Женевы , Швейцария | Университет Лозанны , Швейцария | Университет Лозанны , Швейцария |
| Калифорнийский Университет , Беркли, США | Университет Санта Круз ( UCSC ), Калифорния, США | Университет Санта Круз ( UCSC ), Калифорния, США |
| Европейский Биоинформатический Институт, Хинкстон, Великобритания | Массачусетский технологический институт (MIT), Бостон США | Массачусетский технологический институт (MIT), Бостон, США |
| Йельский университет , Нью-Хейвен, США | Йельский университет , Нью-Хейвен, США | |
| , Мадрид, Испания | , Мадрид, Испания | |
| Университет Вашингтона ( WashU ), Сент-Луис, США | Европейский Биоинформатический Институт , Кэмбридж, Великобритания |
Основная статистика
Полнота данных в аннотациях GENCODE непрерывно растёт. Ниже приведена статистика версии GENCODE 28 . Эта версия соответствует выпуску Ensembl 92 и содержит аннотацию, сделанную по сборке человеческого генома , но доступную также для сборки ).
| Категории | Всего | Категории | Всего |
|---|---|---|---|
| Общее количество генов | 58 381 | Всего транскриптов | 203 835 |
| Белок-кодирующие гены | 19 901 | Белок-кодирующие транскрипты: | 82 335 |
| Гены длинных некодирующих РНК | 15 779 | - кодирующие полный белок | 56 541 |
| Гены малых некодирующих РНК | 7 569 | - кодирующие фрагмент белка | 25 794 |
| Псевдогены : | 14 723 | Нонсенс-опосредованно распавшиеся транскрипты | 14 889 |
| - обработанные псевдогены | 10 693 | Транскрипты локусов длинных некодирующих РНК | 28 468 |
| - необработанные псевдогены | 3 519 | ||
| - унитарные псевдогены | 218 | ||
| - полиморфные псевдогены | 38 | ||
| - псевдогены | 18 | ||
| Генные сегменты иммуноглобулин-T-клеточного рецептора : | 645 | Общее число различных аннотаций | 61 132 |
| - белок-кодирующие сегменты | 408 | Число генов, к которым относится больше одной аннотации | 13 641 |
| - псевдогены | 237 |
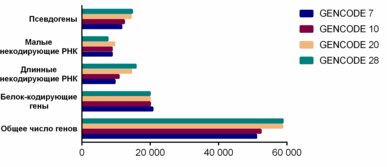
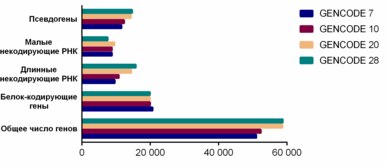
Сравнение версий GENCODE
Сравнительная статистика четырёх основных релизов GENCODE продемонстрирована на графике справа .
Данные свидетельствуют о том, что покрытие генома (количество обнаруженных и аннотированных локусов) стабильно увеличивается. При этом доля белок-кодирующих генов среди аннотированных уменьшается — в основном, из-за результатов аннотирования поли(A) -сайтов и кэп -анализа экспрессии генов ( CAGE ) . Одновременно с этим увеличивается количество псевдогенов и локусов, аннотированных как длинные некодирующие РНК .
Методология
В проекте GENCODE применялись ручная и автоматическая аннотация. При верификации результатов использовались данные лабораторных экспериментов .
Автоматическая аннотация (ENSEMBL)
Информация о транскриптах Ensembl , полученных при автоматической аннотации генов, основывалась на экспериментальных данных о последовательностях белков и мРНК из публичных баз данных . Помимо белок-кодирующих участков, аннотировались нетранслируемые участки, длинные некодирующие РНК и короткие некодирующие РНК .

Аннотация вручную (группа HAVANA)
HAVANA (Human and Vertebrate Analysis and Annotation) — исследовательская группа, осуществляющая ручную аннотацию транскриптов в проекте GENCODE .
Помимо группы HAVANA, в состав консорциума GENCODE входило ещё несколько групп, проводивших анализ аннотированных локусов с помощью программ Ensembl и помогавших аннотаторам в идентификации пропущенных либо неверно аннотированных локусов, в том числе сайтов сплайсинга . Для обмена информацией между группами использовалась трекинговая система AnnoTrack . В процессе также использовались данные экспериментов РНК-секвенирования , CAGE и Ditag .
Помимо официально вовлечённых в проект групп, над улучшением качества аннотации GENCODE работали независимые исследователи .
Объединение ручной и автоматической аннотаций
При объединении все модели транскриптов, полученные автоматической и ручной аннотацией, сравнивались для перекрывающихся транскриптов. Найденные расхождения детектировались с использованием системы AnnoTrack .
Автоматическая аннотация не всегда может считаться корректной (так, только в 45 % случаев автоматической аннотации корректно предсказываются все альтернативные транскрипты ). В случае несовпадения аннотаций приоритетной являлась аннотация HAVANA, так как ручная аннотация в сложных случаях предполагала анализ геномного контекста, литературы и использование экспериментальных данных Mus musculus . Тем не менее, для таких случаев сохраняется информация, полученная обоими способами аннотации .
Оценка качества
Транскриптам присваивается определённый уровень поддержки на основании сравнения транскрипта GENCODE с выравниванием мРНК и маркерных экспрессирующихся последовательностей (EST), полученным из Ensembl и UCSC. В итоге аннотации каждого транскрипта делятся на определённые вручную, автоматически или слитые аннотации, то есть те, для которых и автоматический метод, и метод аннотирования вручную дали одинаковые результаты .
Основные подходы, использованные в GENCODE 7
Амплификация, секвенирование, картирование и валидация результатов
С помощью амплификации кДНК были сгенерированы двуцепочечные кДНК человеческих тканей ( мозга , сердца , почки , яичка , печени , селезёнки , лёгкого и скелетной мышцы ). Очищенная ДНК использовалась для создания геномной библиотеки с помощью набора Illumina «Genomic DNA sample prep kit». Библиотека была отсеквенирована на платформе Illumina Genome Analyzer 2. (по 35 или 75 нуклеотидов ) картировались на человека сборки и предсказанные с использованием программы Bowtie. Транскрипты валидировались только ридами, которые однозначно картировались на геном. Места соединений экзонов считались достоверными, если на них картировалось минимум 10 ридов, содержащими хотя бы 4 (для ридов длиной 35 нуклеотидов) или 8 (для ридов длиной 75 нуклеотидов) нуклеотидов в каждом из двух экзонов, разделённых сайтом сплайсинга .
Прочие подходы
Для аннотации альтернативных транскриптов генов использовался веб-сервис . APPRIS выбирает один вариант в качестве «главной изоформы» на основании информации о белковом продукте гена и об ортологах близких видов. APPRIS широко использовался при масштабировании проекта ENCODE и при аннотации геномов других видов ( Mus sp. , Danio sp. , Rattus sp. ) .
Для поиска кодирующих последовательностей в транскриптах, автоматически предсказанных на основании данных РНК-секвенирования , использовалась программа PhyloCSF. Она основывается на анализе паттернов эволюции , выравнивая транскрипт с экзонами позвоночных из UCSC (включая 33 плацентарных млекопитающих) .
Организация данных
Текущая версия набора генов человека в GENCODE включает файлы аннотаций (в форматах GTF и GFF3 ), FASTA -файлы и файлы METADATA, связанные с аннотацией GENCODE для всех геномных участков . Они соотнесены с референсной хромосомой и хранятся в отдельных файлах, которые содержат: генную аннотацию, сайты полиаденилирования , аннотированные группой HAVANA, псевдогены, предсказанные алгоритмами Йельского университета и университета Санта-Круз (США) , длинные некодирующие РНК, а также структуры тРНК , предсказанные tRNA-Scan .
Определение уровня аннотации
Все гены датасета GENCODE классифицируются на три категории в соответствии с типом аннотации :
- Уровень 1 (подтвержденный локус): включает транскрипты, которые были аннотированы вручную и проверены экспериментально с помощью RT-PCR — секвенирования, а также псевдогены, подтвержденные тремя разными методологиями .
- Уровень 2 (аннотированный вручную локус): к нему относятся транскрипты, аннотированые только вручную группой HAVANA, а также транскрипты, совмещенные с моделями, полученными по автоматическому протоколу Ensembl .
- Уровень 3 (автоматически аннотированный локус): отражает транскрипты или псевдогены, предсказанные только с помощью автоматической аннотации Ensembl .
Определение статуса гена/транскрипта
Генам и транскриптам присваиваются статусы «известный», «новый» и «предполагаемый» в зависимости от их представленности в других основных базах данных и от оснований, использованных для построения составляющих их транскриптов .
Известный («known»): представлен в базах данных (HGNC) и .
Новый («novel»): не представлен в базах HGNC или RefSeq, но хорошо подтверждается либо транскриптом, специфичным для данного локуса, либо свидетельствами его присутствия в паралогичном или ортологичном локусе .
Предполагаемый («putative»): не представлен в базах НGNC или RefSeq, но подтверждается признаками существования более короткого и редкого транскрипта .
Связанные с GENCODE проекты
Ensembl
Проект Ensembl является важной частью проекта ENCODE и представляет собой геномный браузер, позволяющий визуализировать сборку генома и все данные проекта ENCODE, в частности, аннотированные в проекте GENCODE геномные участки .
RGASP
RGASP (The RNA-seq Genome Annotation Assessment Project) — проект, организованный в рамках консорциума GENCODE после семинара EGASP (ENCODE Genome Annotation Assessment Project) по предсказанию генов. Было проведено две сессии семинаров для анализа результатов секвенирования РНК , а также рассмотрения его различных (методических и технических) аспектов. Одной из наиболее существенных находок первых двух стадий проекта стала важность соотнесения чтения с качеством полученного предсказания гена. В 2014 году была проведена третья сессия семинаров RGASP, где основное внимание было уделено картированию чтений на геном. Проект предоставил софт для аннотации транскриптов (определение, реконструкция и расчет количества транскриптов) .
Примечания
- Williams F. M. , Scollen S. , Cao D. , Memari Y. , Hyde C. L. , Zhang B. , Sidders B. , Ziemek D. , Shi Y. , Harris J. , Harrow I. , Dougherty B. , Malarstig A. , McEwen R. , Stephens J. C. , Patel K. , Menni C. , Shin S. Y. , Hodgkiss D. , Surdulescu G. , He W. , Jin X. , McMahon S. B. , Soranzo N. , John S. , Wang J. , Spector T. D. (англ.) // PLoS Genetics. — 2012. — Vol. 8 , no. 12 . — P. e1003095—1003095 . — doi : . — .
- (англ.) . ENCODE . Stanford University. — Официальный сайт проекта и одноименного консорциума ENCODE. Дата обращения: 19 мая 2018. 28 марта 2021 года.
- ↑ Harrow J. , Denoeud F. , Frankish A. , Reymond A. , Chen C. K. , Chrast J. , Lagarde J. , Gilbert J. G. , Storey R. , Swarbreck D. , Rossier C. , Ucla C. , Hubbard T. , Antonarakis S. E. , Guigo R. (англ.) // Genome Biology. — 2006. — Vol. 7 Suppl 1 . — P. 4—1 . — doi : . — .
- ↑ Harrow J. , Frankish A. , Gonzalez J. M. , Tapanari E. , Diekhans M. , Kokocinski F. , Aken B. L. , Barrell D. , Zadissa A. , Searle S. , Barnes I. , Bignell A. , Boychenko V. , Hunt T. , Kay M. , Mukherjee G. , Rajan J. , Despacio-Reyes G. , Saunders G. , Steward C. , Harte R. , Lin M. , Howald C. , Tanzer A. , Derrien T. , Chrast J. , Walters N. , Balasubramanian S. , Pei B. , Tress M. , Rodriguez J. M. , Ezkurdia I. , van Baren J. , Brent M. , Haussler D. , Kellis M. , Valencia A. , Reymond A. , Gerstein M. , Guigó R. , Hubbard T. J. (англ.) // Genome Research. — 2012. — September ( vol. 22 , no. 9 ). — P. 1760—1774 . — doi : . — .
- Frankish A. , Mudge J. M. , Thomas M. , Harrow J. (англ.) // Database : The Journal Of Biological Databases And Curation. — 2012. — Vol. 2012 . — P. 014—014 . — doi : . — .
- ↑ Derrien T. , Johnson R. , Bussotti G. , Tanzer A. , Djebali S. , Tilgner H. , Guernec G. , Martin D. , Merkel A. , Knowles D. G. , Lagarde J. , Veeravalli L. , Ruan X. , Ruan Y. , Lassmann T. , Carninci P. , Brown J. B. , Lipovich L. , Gonzalez J. M. , Thomas M. , Davis C. A. , Shiekhattar R. , Gingeras T. R. , Hubbard T. J. , Notredame C. , Harrow J. , Guigó R. (англ.) // Genome Research. — 2012. — September ( vol. 22 , no. 9 ). — P. 1775—1789 . — doi : . — .
- Pei B. , Sisu C. , Frankish A. , Howald C. , Habegger L. , Mu X. J. , Harte R. , Balasubramanian S. , Tanzer A. , Diekhans M. , Reymond A. , Hubbard T. J. , Harrow J. , Gerstein M. B. (англ.) // Genome Biology. — 2012. — 26 September ( vol. 13 , no. 9 ). — P. 51—51 . — doi : . — .
- (англ.) . National Human Genome Research Institute (1 октября 2015). — О проекте "Геном человека". Дата обращения: 12 мая 2018. 2 мая 2018 года.
- ↑ ENCODE Project Consortium. (англ.) // Science (New York, N.Y.). — 2004. — 22 October ( vol. 306 , no. 5696 ). — P. 636—640 . — doi : . — .
- ENCODE Project Consortium, Ewan Birney, John A. Stamatoyannopoulos, Anindya Dutta, Roderic Guigó. // Nature. — 2007-06-14. — Т. 447 , вып. 7146 . — С. 799–816 . — ISSN . — doi : . 19 июня 2019 года.
- ↑ (англ.) . Wellcome Trust Sanger Institute. — Описание проекта GENCODE на официальном сайте проекта. Дата обращения: 12 мая 2018. Архивировано из 29 апреля 2018 года.
- ↑ (англ.) . Wellcome Trust Sanger Institute. — Выпуск GENCODE 28 (апрель 2018). Дата обращения: 12 мая 2018. Архивировано из 12 апреля 2018 года.
- ↑ Mark B. Gerstein, Can Bruce, Joel S. Rozowsky, Deyou Zheng, Jiang Du. // Genome Research. — June 2007. — Т. 17 , вып. 6 . — С. 669–681 . — ISSN . — doi : . 20 июня 2018 года.
- E. F. Vanin. // Annual Review of Genetics. — 1985. — Т. 19 . — С. 253–272 . — ISSN . — doi : . 13 мая 2018 года.
- Jinrui Xu, Jianzhi Zhang. (англ.) // Molecular Biology and Evolution. — 2016-03-01. — Vol. 33 , iss. 3 . — P. 755–760 . — ISSN . — doi : . 14 мая 2018 года.
- (англ.) . GENCODE . Wellcome Trust Sanger Institute. — Дизайн микрочипов экспрессии длинных некодирующих РНК для проекта GENCODE. Дата обращения: 13 мая 2018. Архивировано из 8 апреля 2018 года.
- Igor Ulitsky, David P. Bartel. // Cell. — 2013-07-03. — Т. 154 , вып. 1 . — С. 26–46 . — ISSN . — doi : . 21 августа 2017 года.
- (англ.) . Wellcome Sanger Institute. — Список участников проекта GENCODE на официальном сайте проекта. Дата обращения: 13 мая 2018. Архивировано из 11 мая 2018 года.
- (англ.) . GENCODE . Wellcome Sanger Institute (апрель 2018). — Все выпуски аннотаций человеческого генома на официальном сайте GENCODE. Дата обращения: 13 мая 2018. Архивировано из 14 апреля 2018 года.
- (англ.) . GENCODE . Wellcome Trust Sanger Institute. — Статистика выпуска GENCODE 21. Дата обращения: 13 мая 2018. Архивировано 8 апреля 2018 года.
- (англ.) . GENCODE . Wellcome Trust Sanger Institute. — Описание задач проекта GENCODE на официальном сайте проекта. Дата обращения: 13 мая 2018. 8 апреля 2018 года.
- (англ.) . Archive!Ensembl . EMBL-EBI. — Описание данных Ensembl на официальном сайте проекта. Дата обращения: 13 мая 2018.
- Mudge J. M. , Harrow J. (англ.) // Mammalian Genome : Official Journal Of The International Mammalian Genome Society. — 2015. — October ( vol. 26 , no. 9-10 ). — P. 366—378 . — doi : . — .
- Kokocinski F. , Harrow J. , Hubbard T. (англ.) // BMC Genomics. — 2010. — 5 October ( vol. 11 ). — P. 538—538 . — doi : . — .
- S. Searle, A. Frankish, A. Bignell, B. Aken, T. Derrien. // Genome Biology. — 2010-10-11. — Т. 11 , вып. 1 . — С. P36 . — ISSN . — doi : .
- Wright J. C. , Mudge J. , Weisser H. , Barzine M. P. , Gonzalez J. M. , Brazma A. , Choudhary J. S. , Harrow J. (англ.) // Nature Communications. — 2016. — 2 June ( vol. 7 ). — P. 11778—11778 . — doi : . — .
- Jose Manuel Rodriguez, Juan Rodriguez-Rivas, Tomás Di Domenico, Jesús Vázquez, Alfonso Valencia. (англ.) // Nucleic Acids Research. — 2017-10-23. — Vol. 46 , iss. D1 . — P. D213–D217 . — ISSN . — doi : . 13 мая 2018 года.
- (англ.) . Ensembl . EMBL-EBI. — Описание использования данных проекта ENCODE на сайте Ensembl. Дата обращения: 12 мая 2018. 9 ноября 2017 года.
- . RNA-seq Genome Annotation Assessment Project (англ.) . GENCODE . Wellcome Sanger Institute. — Описание проекта RGASP на официальном сайте GENCODE. Дата обращения: 13 мая 2018. Архивировано из 8 апреля 2018 года.
Ссылки
- 2020-07-07
- 1
