Interested Article - Альтернативный сплайсинг

- 2020-06-04
- 1

Альтернати́вный спла́йсинг — вариант сплайсинга матричных РНК (мРНК), при котором в ходе экспрессии гена на основе одного и того же первичного транскрипта (пре-мРНК) происходит образование нескольких зрелых мРНК. Структурные и функциональные различия образовавшихся транскриптов могут быть вызваны как выборочным включением в зрелую мРНК экзонов первичного транскрипта, так и сохранением в ней частей интронов . Наиболее распространённая разновидность альтернативного сплайсинга предусматривает : отдельные экзоны транскрипта при определённых условиях могут быть как включены в зрелую мРНК, так и пропущены .
Белки , получаемые трансляцией таких мРНК, в результате имеют разные аминокислотные последовательности; таким образом, при альтернативном сплайсинге один транскрипт обеспечивает синтез нескольких белков. Широкое распространение такого сплайсинга у эукариот приводит к значительному увеличению разнообразия белков, закодированных в их геномах . Например, организм человека синтезирует не менее чем 100 тысяч различных белков, в то время как число кодирующих их генов примерно 20 тысяч (при этом среди всех генов человека, которые содержат интроны, более 75 % выступают как матрицы для синтеза пре-мРНК, подвергаемых далее альтернативному сплайсингу) .
Образование альтернативно сплайсированных мРНК находится под контролем системы транс -действующих белков ( ), которые связываются с цис -сайтами первичного транскрипта. Среди факторов сплайсинга выделяют активаторы и репрессоры сплайсинга: первые способствуют использованию отдельных его сайтов, а вторые, наоборот, предотвращают их использование. Механизмы альтернативного сплайсинга очень разнообразны, знание «кода сплайсинга» создаёт возможность предсказывать результаты сплайсинга конкретного гена в тех или иных условиях .
Аномалии альтернативного сплайсинга нередко приводят к болезням; немало генетических заболеваний человека вызвано этими аномалиями . Исследователи полагают, что аберрантный сплайсинг может способствовать развитию рака , причём показано, что при различных видах рака гены факторов сплайсинга часто мутируют , приводя к нарушению нормального хода сплайсинга . Установлено также, что аномалии альтернативного сплайсинга вносят вклад в развитие резистентности организма к химиотерапии .
История изучения
Впервые альтернативный сплайсинг был описан в 1977 году у аденовирусов . Было установлено, что аденовирус образует пять различных транскриптов в ранней стадии инфекционного цикла, до репликации вирусной ДНК , и ещё один после начала репликации ДНК; при этом образование ранних первичных транскриптов продолжается после начала репликации ДНК. Дополнительный одиночный транскрипт, образуемый на поздних стадиях инфекционного цикла, считывается с 5/6 аденовирусного генома размером 32 килобазы. Поздний транскрипт гораздо длиннее каждого из ранних вирусных транскриптов. Исследователи показали, что первичный транскрипт, образуемый аденовирусом типа 2 на поздних стадиях инфекции, подвергается сплайсингу разными способами, что приводит к образованию мРНК, кодирующих разные вирусные белки. Кроме того, первичный транскрипт содержит множество сайтов полиаденилирования , в результате чего у разных мРНК могут получаться разные 3'-концы .
В 1981 году альтернативный сплайсинг был описан у клеточного эукариотического гена. Было показано, что в клетках млекопитающих такой альтернативный сопровождает образование гормона кальцитонина . Первичный транскрипт гена кальцитонина содержит 6 экзонов; в зрелую мРНК, кодирующую кальцитонин, входят экзоны 1—4, и сигнал полиаденилирования находится в экзоне 4. У другой мРНК, образуемой из того же первичного транскрипта, при сплайсинге экзон 4 пропускается, и зрелая мРНК содержит экзоны 1—3, 5 и 6. Она кодирует белок, известный как ( англ. calcitonin gene related peptide ) . В начале 1980-х годов был также открыт альтернативный сплайсинг в генах иммуноглобулинов млекопитающих .
Последующие исследования показали, что альтернативный сплайсинг распространён среди всех эукариот . При этом количество изоформ белка, которые могут быть транслированы с одного гена, может быть весьма значительным. Так, подсчитано, что ген плодовой мушки Drosophila melanogaster , известный как , при независимом комбинировании в мРНК всех имеющихся экзонов потенциально может обеспечить синтез 38 016 изоформ .
Модели

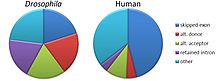
Существует пять моделей альтернативного сплайсинга :
- Пропуск экзона или кассетный экзон . В данном случае любой экзон может быть вырезан или включён в состав зрелой мРНК. Это — наиболее распространённая модель прохождения альтернативного сплайсинга у пре-мРНК млекопитающих, по некоторым подсчётам, более 38 % событий альтернативного сплайсинга у млекопитающих следует данной модели ).
- Взаимоисключающие экзоны . Из двух экзонов в зрелую мРНК включается только один, но не оба (данная модель сложнее остальных, поскольку предусматривает скоординированное протекание, по крайней мере, двух событий альтернативного сплайсинга ).
- Альтернативный донорный сайт . Используется альтернативный 5'-конец интрона (донорный сайт), так что меняется 3'-конец вышестоящего экзона (у млекопитающих данный механизм ответственен примерно за 8 % событий альтернативного сплайсинга ).
- Альтернативный акцепторный сайт . Используется альтернативный 3'-конец интрона (акцепторный сайт), так что меняется 5'-конец нижестоящего экзона (на этой модели основано примерно 18 % событий альтернативного сплайсинга у млекопитающих ).
- Удержание интрона . Последовательность может быть вырезана как интрон или оставлена в зрелой мРНК. Этот способ отличается от пропуска экзона, поскольку сохраняемая последовательность не окружена интронами. Если оставленный интрон попадает в кодирующую область , то он должен кодировать аминокислотную последовательность с такой же рамкой считывания , как и соседние экзоны. Если же он будет иметь другую рамку считывания или содержать стоп-кодон , белок будет нефункциональным. Это — самый редкий механизм альтернативного сплайсинга у млекопитающих (на него приходится примерно 3 % событий АС; в то же время, данный механизм достаточно широко распространён при онкологических заболеваниях, где он служит одним из механизмов инактивации генов-супрессоров опухолей ) .
Кроме пяти основных моделей альтернативного сплайсинга, известны два способа получения нескольких белков с одного гена в результате использования множественных промоторов и множественных сайтов полиаденилирования . Однако, использование множественных промоторов относится, скорее, к регуляции транскрипции, чем к альтернативному сплайсингу. Начиная транскрипцию с разных точек, можно получить транскрипты с различающимися 5'-концевыми экзонами. С другой стороны, использование множественных сайтов полиаденилирования приводит к образованию разных 3'-концов у созревающих транскриптов. Оба этих механизма в сочетании с пятью моделями сплайсинга обеспечивают разнообразие мРНК, считываемых с одного и того же гена .

Один транскрипт способен подвергаться более чем одному типу альтернативного сплайсинга . Рассмотренные выше модели хорошо описывают базовые механизмы сплайсинга, однако могут не подходить для сложных случаев. Например, на рисунке справа представлены три формы гена гиалуронидазы 3 мыши, полученные в ходе сплайсинга. Сравнение экзонов первой формы (зелёная) и второй (жёлтая) указывает, что интрон был сохранён в конечном транскрипте, а сравнение второй формы с третьей (синяя) показывает пропуск экзона .
Механизм
Общая схема сплайсинга
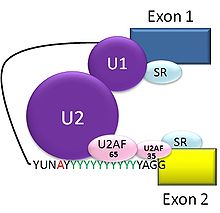
Пре-мРНК, транскрибируемая с ДНК, содержит как экзоны, так и интроны, причём число и длина интронов, создающих необходимый фон для альтернативного сплайсинга, у различных эукариот существенно варьирует. Так, среднее число интронов, приходящееся на один интрон-содержащий ген, у модельных организмов составляет: у дрозофилы Drosophila melanogaster — 2,5, у нематоды Caenorhabditis elegans — 4,2, у резуховидки Таля Arabidopsis thaliana — 4,8; у млекопитающих оно изменяется от 5,7 до 7,8 . В ходе сплайсинга экзоны должны быть оставлены в транскрипте, а интроны удалены. Регуляцию и выбор сайтов сплайсинга обеспечивают транс -действующие белки-активаторы и репрессоры сплайсинга, а также присутствующие в самой пре-мРНК цис -действующие элементы — энхансеры и сайленсеры сплайсинга .
Типичные эукариотические интроны содержат консенсусные последовательности ; так, на 5'-конце каждого интрона присутствует ди нуклеотид GU, рядом с 3'-концом находится «точка ветвления», в которой всегда присутствует нуклеотид А , а расположенные вокруг него последовательности варьируются. У человека консенсусная последовательность вокруг точки ветвления yUnAy . После точки ветвления расположен ряд пиримидинов ( ), а 3'-конец интрона выглядит как AG .
Сплайсинг пре-мРНК осуществляет РНК -белковый комплекс, известный как сплайсосома . В состав сплайсосомы входят (snRNP), обозначаемые , , , и (рибонуклеотид U3 в сплайсинге мРНК не участвует) . Рибонуклеотид U1 связывается с 5'-концевым динуклеотидом GU, а U2, при участии белковых факторов U2AF, связывается с точкой ветвления (на этой стадии комплекс называется сплайсосомный А-комплекс). Во время формирования А-комплекса определяются 5'- и 3'-границы удаляемого интрона, а также концы экзонов, которые должны быть оставлены .
Далее с А-комплексом связывается комплекс U4, U5, U6. После этого U6 замещает U1, а U1 и U4 покидают комплекс. Оставшийся комплекс подвергается двум реакциям переэтерификации . В ходе первой реакции происходит отрезание 5'-конца интрона от вышележащего экзона и его присоединение в точке ветвления к нуклеотиду А с помощью 2',5'- , в результате чего интрон принимает форму лассо . Вторая реакция обеспечивает отрезание 3'-конца интрона и соединение двух экзонов фосфодиэфирной связью; при этом интрон высвобождается и разрушается .
Регуляторные элементы и белки

Сплайсинг регулируется транс -действующими белками ( активаторами и репрессорами ) и соответствующими цис -регуляторными элементами ( сайленсерами и энхансерами ) на пре-мРНК. Впрочем, имеются данные, что во многих случаях действие фактора сплайсинга зависит от его положения: когда фактор сплайсинга связан с интронным энхансерным элементом, он действует как активатор сплайсинга, а когда связывается с регуляторным сайтом в экзоне, то действует как репрессор . В регуляции сплайсинга принимает также участие вторичная структура пре-мРНК, которая обеспечивает эффективное сближение друг с другом двух регуляторных элементов или маскирует те последовательности, которые могли бы служить местами связывания факторов сплайсинга . Вместе все эти элементы образуют «код сплайсинга», который определяет, как будет проходить сплайсинг в данных клеточных условиях .
Известны два типа цис -активирующих элементов в пре-мРНК, и им соответствуют транс -активирующие РНК-связывающие белки. Сайленсеры сплайсинга — это элементы, с которыми связываются белки-репрессоры сайленсинга, снижая при этом вероятность того, что по соседству будет находиться сайт сплайсинга. Местом размещения сайленсеров сплайсинга могут быть как интроны (интронные сайленсеры сплайсинга, ISS), так и экзоны (экзонные сайленсеры сплайсинга, ESS). Их нуклеотидные последовательности, как и связывающиеся с ними белки, весьма разнообразны. Большинство репрессоров сплайсинга являются (hnRNP) — такими, как и белок, связывающий полипиримидиновый тракт (PTB) .
С энхансерами сплайсинга связываются белки-активаторы сплайсинга, увеличивая эффективную вероятность того, что рядом будет находиться сайт сплайсинга. Их местом размещения также могут служить как интроны (интронные энхансеры сплайсинга, ISE), так и экзоны (экзонные энхансеры сплайсинга, ESE). Бо́льшая часть белков, связывающихся с ISE и ESE, относится к семейству белков (регулирующим не только ход альтернативного сплайсинга, но и многие другие клеточные процессы ; первый из белков данного семейства, идентифицированный как фактор сплайсинга, был открыт в 1991 году ). Эти белки содержат мотивы , распознающие РНК, а также домены , обогащённые аргинином и серином .

Таким образом, факторы сплайсинга действуют взаимозависимо, причём результаты их действия зависят также от окружения . Наличие определённых цис -регуляторных последовательностей РНК способно как увеличить вероятность того, что рядом будет находиться сайт сплайсинга, так и уменьшить эту вероятность — в зависимости от контекста. Например, некоторые такие элементы влияют на сплайсинг только при наличии рядом с ними других вполне определённых элементов. Кроме того, цис -регуляторные элементы могут давать разный эффект при экспрессии в клетке определённых белков. Адаптивное значение энхансеров и сайленсеров сплайсинга подтверждают работы, показывающие, что в человеческих генах мутации, приводящие к образованию новых сайленсеров или разрушению старых энхансеров, подвержены строгому отбору .
Примеры
Пропуск экзона: ген dsx дрозофилы

Пре-мРНК гена дрозофилы D. melanogaster содержит 6 экзонов. У самцов в зрелую мРНК входят экзоны 1, 2, 3, 5, 6, и они кодируют белок, который функционирует как регулятор транскрипции в развитии по мужскому типу. У самок в зрелую мРНК входят экзоны 1, 2, 3 и 4, причём экзон 4 содержит сигнал полиаденилирования, по которому мРНК разрезается. Получившийся белок функционирует как регулятор транскрипции в развитии по женскому типу .
В описанном примере имеет место альтернативный сплайсинг по типу пропуска экзона. Интрон, лежащий выше экзона 4, содержит полипиримидиновый тракт, который не вполне удовлетворяет консенсусной последовательности сплайсинга, поэтому белки U2AF в отсутствие активаторов сплайсинга связываются с ним плохо. По этой причине у самцов этот 3'-акцепторный сайт сплайсинга не используется. У самок, однако, присутствует активатор сплайсинга Transformer (Tra). Этот белок связывается с SR-белком Tra2 (который образуется у обоих полов и связывается с ESE в экзоне 4) и совместно с ещё одним SR-белком, dsxRE, формирует комплекс, который содействует связыванию белков U2AF со слабым пиримидиновым трактом. U2 рекрутируется к соответствующей точке ветвления, и в результате происходит включение экзона 4 в состав зрелой мРНК .
Альтернативные акцепторные сайты: Transformer дрозофилы

Пре-мРНК гена Transformer (Tra) D. melanogaster подвергаются альтернативному сплайсингу по модели альтернативных акцепторных сайтов. Ген Tra кодирует белок, экспрессия которого имеет место только у самок. Первичный транскрипт этого гена содержит интрон с двумя возможными акцепторными сайтами. У самцов задействован вышележащий акцепторный сайт, благодаря чему происходит включение в мРНК удлинённого варианта экзона 2, содержащего преждевременный стоп-кодон; поэтому у самцов образуется укороченный неактивный белок. У самок же образуется полноценный белок, играющий ключевую роль в определении пола и известный как Sex lethal (Sxl). Белок Sxl является репрессором сплайсинга и, связываясь с ISS в РНК-транскрипте Tra рядом с вышележащим акцепторным сайтом, предотвращает связывание белка U2AF с полипиримидиновым трактом; в результате сплайсосома связывается с нижележащим акцепторным сайтом, что приводит к удалению преждевременного стоп-кодона. Полученная мРНК кодирует белок Tra, который сам выступает регулятором альтернативного сплайсинга других связанных с полом генов (см. выше пример гена dsx ) .
Альтернативный сплайсинг рецептора Fas

В результате альтернативного сплайсинга происходит синтез множества изоформ рецептора . У человека две нормальные изоформы данного рецептора образуются по механизму пропуска экзона. мРНК, содержащая 6 экзонов, кодирует мембраносвязанную форму рецептора Fas, которая стимулирует апоптоз . Повышенное образование рецептора Fas в клетках, постоянно подвергающихся воздействию солнечного света, и отсутствие этого рецептора в клетках рака кожи свидетельствуют о том, что рассматриваемый механизм играет важную роль в элиминации клеток, вставших на путь превращения в раковые . При пропуске экзона 6 образуется водорастворимая изоформа белка Fas, которая стимулировать апоптоз неспособна. Выбор между вставкой или пропуском экзона зависит от действия двух белков-антагонистов: и PTB.
Донорный сайт, расположенный на 5'-конце интрона, следующим после экзона 6 в пре-мРНК, плохо согласуется с консенсусной последовательностью сплайсинга, и не всегда связывается с snRNP U1 . Если связывания U1 не происходит, экзон 6 пропускается (картинка а на рисунке справа). Связывание белка TIA-1 с интронным энхансером сплайсинга стабилизирует связывание U1. Образующийся на 5'-конце интрона донорный сайт помогает связываться фактору сплайсинга U2AF с 3'-сайтом сплайсинга, расположенным выше экзона, хотя механизм этого ещё не понятен (картинка b на рисунке справа) . Экзон 6 содержит обогащённый пиримидинами сайленсер сплайсинга ( ure6 ), с которым может связываться PTB. Если связывание PTB имеет место, то донорный сайт на 5'-конце интрона не способствует связыванию фактора U2AF, и экзон пропускается (картинка с на рисунке справа).
Описанный выше механизм является примером определения экзона при сплайсинге. Сплайсосома собирается в области интрона, и snRNP укладывают РНК так, что 5'- и 3'-концы интрона соединяются. Однако в описанном выше случае происходит также взаимодействие концов экзона. В этом случае взаимодействия, определяющие границы экзона, необходимы для связывания коровых факторов сплайсинга до сборки сплайсосомы на границах фланкирующих интронов .
Конкуренция репрессора и активатора: экзон 2 гена tat ВИЧ-1

ВИЧ — ретровирус , являющийся причиной развития СПИДа — образует единственную пре-мРНК, из которой далее посредством альтернативного сплайсинга образуется более 40 различных мРНК . Равновесие между сплайсированными по-разному транскриптами обеспечивает образование мРНК, кодирующих все белки, необходимые для репликации вируса . Один из по-разному сплайсированных транскриптов содержит транскрипт гена , у которого экзон 2 является кассетным, то есть может быть либо включён в итоговый транскрипт, либо не включён. Включение этого экзона регулируют репрессор сплайсинга hnRNP A1 и SR-белок SC35. В экзоне 2 имеет место перекрывание сайленсерной последовательности (ESS) и энхансерной последовательности (ESE) перекрываются. Если с ESS связывается репрессор А1, то он запускает кооперативное связывание молекул А1, закрывая 5'-концевой донорный сайт выше экзона 2 и препятствуя связыванию U2AF35 с полипиримидиновым трактом. Если SC35 связывается с ESE, то он препятствует связыванию А1, и 5'-донорный сайт остаётся доступным для сплайсосомы. Конкуренция между репрессором и активатором приводит к образованию РНК, соответственно содержащей или не содержащей экзон 2 .
Адаптивное значение
Альтернативный сплайсинг — это одно из исключений из правила, по которому одному гену соответствует один белок (гипотеза «один ген — один фермент ») . Корректнее было бы сказать: «один ген — много полипептидов ». Внешняя информация нужна для того, чтобы решить, какой именно полипептид образовать с данной мРНК. Поскольку способы регуляции наследуются, то это открывает мутациям новый путь к изменению экспрессии генов .
Предполагается, что для эукариот альтернативный сплайсинг — очень важный шаг на пути к повышению эффективности экспрессии генов, поскольку он даёт возможность хранить информацию более экономно. Один ген может давать начало нескольким белкам, а не одному, поэтому одно и то же разнообразие протеома можно получить с генома существенно меньшего размера . Это также обеспечивает эволюционную гибкость. Единственная точечная мутация может привести к включению или исключению экзона из транскрипта, благодаря чему может быть получена новая изоформа белка без потери его основной формы . Обнаружены действительно неупорядоченные регионы, которые содержат много неконститутивных экзонов, поэтому изоформы белка могут выполнять новые функции, изменяя функциональные модули в этих местах . Сравнительные оценки показывают, что возникновение альтернативного сплайсинга в ходе эволюции предшествовало появлению многоклеточности; предполагают, что альтернативный сплайсинг был одним из средств, обеспечивающих возникновение многоклеточных организмов .
Исследования в рамках проекта «Геном человека» , а также других проектов по секвенированию геномов показали, что геном человека всего лишь на 30 % больше генома нематоды Caenorhabditis elegans и всего лишь в два раза больше, чем у плодовой мушки Drosophila melanogaster . Эти данные наводят на мысль, что сложность человека и позвоночных животных вообще может быть связана с более активным, по сравнению с беспозвоночными , использованию альтернативного сплайсинга . Однако дальнейшее изучение геномных последовательностей человека, мыши, крысы , коровы , D. melanogaster , C. elegans и растения Arabidopsis thaliana показало, что между человеком и другими эукариотами не наблюдается значительной разницы в использовании альтернативного сплайсинга . Имеются, впрочем, сведения, что полученные данные представляют собой , связанный с неравномерным включением в сравнительный анализ последовательностей комплементарной ДНК , взятых у различных организмов. При сравнении частот использования альтернативного сплайсинга для случайных выборок генов, полученных от сравниваемых организмов, оказалось, что у позвоночных альтернативный сплайсинг всё же встречается чаще, чем у беспозвоночных .
Клиническое значение
Изменения в аппарате процессинга РНК могут приводить к нарушениям сплайсинга многих транскриптов, а однонуклеотидные замены в сайтах сплайсинга или цис -регуляторных сайтах сплайсинга приводят к различиям в сплайсинге одного и того же гена, как и при сплайсинге транскрипта мутировавшего гена. В работе 2005 года было показано, что свыше 60 % мутаций, приводящих к развитию болезней, влияют не на саму кодирующую последовательность, а на сплайсинг . Показано также, что примерно треть наследственных заболеваний связана с нарушениями сплайсинга .
Аномально сплайсированные мРНК встречаются в заметной доле раковых клеток . Анализ RNA-Seq и протеомов показал выраженные различия в экспрессии сплайсинговых изоформ тех белков, которые участвуют в сигнальных путях , связанных с развитием рака . Неизвестно, влияют ли нарушения сплайсинга на развития рака напрямую, или же они являются следствием поломки клеточных процессов в связи с переходом к раковому росту. Отмечено, что при некоторых видах рака — таких, как рак толстой кишки или рак простаты , количество ошибок в сплайсинге у разных пациентов значительно варьировало; данный феномен назвали транскриптомной нестабильностью .
Кроме этого, было показано, что транскриптомная нестабильность связана с пониженной экспрессией генов факторов сплайсинга. Действительно, в общем случае в раковых клетках альтернативный сплайсинг используется меньше, чем у нормальных клеток, причём модели сплайсинга тоже различаются. Так, в раковых клетках сохранение интрона происходит чаще, чем в нормальных клетках, а пропуск экзона — реже. Особенности сплайсинга в раковых клетках могут быть связаны с высокой частотой соматических мутаций в генах факторов сплайсинга, а некоторые особенности могут быть обусловлены изменениями в фосфорилировании транс -регуляторных факторов сплайсинга . Некоторые особенности сплайсинга могут быть связаны с изменением относительного количества его факторов; например, в клетках рака груди наблюдаются повышенные уровни фактора сплайсинга . В одном исследовании было показано, что относительно небольшая доля (383 из 26000) вариантов альтернативного сплайсинга в раковых клетках встречалась значительно чаще, чем в нормальных; отсюда следует, что существует ограниченное количество генов, аберрантный сплайсинг которых ведёт к развитию опухоли . Считается, однако, что губительное действие нарушенного сплайсинга сдерживается особым клеточным посттранскрипционным механизмом контроля — нонсенс-опосредованным распадом .
Примером гена, специфический вариант сплайсинга которого связан у человека с развитием рака, служит один из генов DNMT . Три гена DNMT кодируют ферменты, которые добавляют метильные группы к ДНК, и модификация данных генов часто имеет регуляторные эффекты. Несколько аномально сплайсированных мРНК гена было найдено в опухолях и клетках раковых . Экспрессия двух из этих мРНК вызывала изменения в метилировании ДНК в данных клетках. Клетки с одной ненормальной мРНК росли вдвое быстрее, чем контрольные клетки, поэтому обнаруженные мРНК связывают с развитием рака .
Другим примером может служить протоонкоген Ron ( MST1R ). Важным свойством раковых клеток является их способность мигрировать ( метастазировать ) в нормальные ткани и нарушать их работу. Образование аномально сплайсированной мРНК Ron было связано с повышенными уровнями SF2/ASF в клетках рака груди. Ненормальная изоформа Ron, транслированная с этой мРНК, увеличивала подвижность клеток .
Сверхэкспрессия укороченного варианта белка — ΔFosB — в специфической популяции нейронов прилежащего ядра лежит в основе возникновения и поддержания привыкания к наркотикам и естественному вознаграждению ( англ. natural reward ) .
Недавние исследования показывают на роль структуры хроматина и модификаций гистонов в регуляции альтернативного сплайсинга. Поэтому эпигенетические факторы могут влиять не только на экспрессию генов, но и на их сплайсинг .
Полногеномный анализ
Полногеномный анализ альтернативного сплайсинга — сложная задача. Обычно альтернативно сплайсированные транскрипты обнаруживают при сравнении ( англ. Expressed sequence tag, EST ). Большинство собраны из очень ограниченного числа тканей, поэтому тканеспецифичные транскрипты ранее не были учтены. Однако появились высокопроизводительные методы для изучения сплайсинга — такие, как ДНК-микрочипы и ( англ. deep sequencing ). Эти методы могут быть использованы для поиска полиморфизмов и мутаций, расположенных в тех элементов сплайсинга, которые влияют на связывание белков, или в их ближайшей окрестности. Сочетая данные методы с такими приёмами исследования сплайсинга, как in vitro анализ репортерных генов , можно изучать влияние полиморфизмов и мутаций на сплайсинг пре-мРНК .
При анализе с помощью микрочипов используют фрагменты ДНК, являющиеся отдельными экзонами (такие, как микрочип ) или границами между экзонами. Затем в микрочип добавляют меченую кДНК из интересующей ткани. Эта пробная кДНК комплементарно связывается с фрагментами ДНК, уже находящимися в микрочипе. Благодаря данному методу можно выявить присутствие определённых альтернативно сплайсированных мРНК .
Метод CLIP ( англ. Cross-linking and immunoprecipitation — образование поперечных сшивок и иммунопреципитация ) использует УФ-излучение для образования сшивок между белками и РНК, подвергающихся сплайсингу. Затем транс -действующие регуляторные белки сплайсинга осаждаются при помощи специальных антител . Когда РНК, связанную с белком, изолируют и клонируют, то определяется последовательность РНК, связанная с регуляторным белком . Использование репортерных генов позволяет выявить белки сплайсинга, участвующие в специфичных случаях альтернативного сплайсинга: в зависимости от того, каким образом прошёл сплайсинг, репортёрный ген будет давать начало двум разным флуоресцентным белкам . Данный метод был использован для выделения мутантов с нарушенным сплайсингом и выявления регуляторных белков сплайсинга, инактивированных у этих мутантов .
См. также
Примечания
- ↑ Blencowe B. J. // Cell. — 2006. — Vol. 126, no. 1. — P. 37—47. — doi : .
- ↑ Дымшиц Г. М., Саблина О. В. // Вавиловский журнал генетики и селекции. — 2014. — Т. 18, № 1 . — С. 71—80 . 16 апреля 2017 года.
- ↑ Black D. L. // Annual Review of Biochemistry. — 2003. — Vol. 72. — P. 291—336. — doi : . — .
- ↑ Pan Qun, Shai O., Lee L. J., Frey B. J., Blencowe B. J. // Nature Genetics. — 2008. — Vol. 40, no. 12. — P. 1413—1415. — doi : . — .
- ↑ Matlin A. J., Clark F., Smith C. W. // Nature Reviews. Molecular Cell Biology. — 2005. — Vol. 6, no. . — P. 386—398. — doi : . — .
- ↑ David C. J., Manley J. L. // Genes & Development. — 2008. — Vol. 22, no. 3. — P. 279—285. — doi : . — .
- ↑ Skotheim R. I., Nees M. Alternative Splicing in Cancer: Noise, Functional, or Systematic? // The International Journal of Biochemistry & Cell Biology. — 2007. — Vol. 39, no. 7-8. — P. 1432—1449. — doi : . — .
- ↑ He Chunjiang, Zhou Fang, Zuo Zhixiang, Cheng Hanhua, Zhou Rongjia. // PLoS One . — 2009. — Vol. 4, no. 3. — P. e4732. — doi : . — .
- ↑ Fackenthal J. D., Godley L. A. // Disease Models & Mechanisms. — 2008. — Vol. 1, no. 1. — P. 37—42. — doi : . — .
- ↑ Sveen A., Kilpinen S., Ruusulehto A., Lothe R. A., Skotheim R. I. // Oncogene. — 2016. — Vol. 35, no. 19. — P. 2413—2427. — doi : . — .
- Zhou Jianbiao, Chng Wee-Joo. // Stem Cell Investigation. — 2017. — Vol. 4, no. 2. — P. 6. — doi : . — .
- Chow L. T., Gelinas R. E., Broker T. R., Roberts R. J. // Cell. — 1977. — Vol. 12, no. 1. — P. 1—8. — doi : . — .
- Berget S. M., Moore C., Sharp P. A. // Proc. Nat. Acad. Sci. USA . — 1977. — Vol. 74, no. 8. — P. 3171—3175. — .
- ↑ Leff S. E., Rosenfeld M. G., Evans R. M. // Annual Review of Biochemistry. — 1986. — Vol. 55. — P. 1091—1117. — doi : . — .
- Chow L. T., Broker T. R. // Cell. — 1978. — Vol. 15, no. 2. — P. 497—510. — doi : . — .
- Nevins J. R., Darnell J. E. // Cell. — 1978. — Vol. 15, no. 4. — P. 1477—1493. — doi : . — .
- Rosenfeld M. G., Amara S. G., Roos B. A., Ong E. S., Evans R. M. // Nature . — 1981. — Vol. 290, no. 5801. — P. 63—65. — .
- Rosenfeld M. G., Lin C. R., Amara S. G., Stolarsky L., Roos B. A., Ong E. S., Evans R. M. // Proc. Nat. Acad. Sci. USA . — 1982. — Vol. 79, no. 6. — P. 1717—1721. — .
- Maki R., Roeder W., Traunecker A., Sidman C., Wabl M., Raschke W., Tonegawa S. // Cell. — 1981. — Vol. 24, no. 2. — P. 353—365. — doi : . — .
- Schmucker D., Clemens J. C., Shu Huidy, Worby C. A., Xiao Jian, Muda M., Dixon J. E., Zipursky S. L. // Cell. — 2000. — Vol. 101, no. 6. — P. 671—684. — doi : . — .
- ↑ Sammeth M., Foissac S., Guigó R. // PLOS Computational Biology . — 2008. — Vol. 4, no. 8. — P. e1000147. — doi : . — .
- ↑ , с. 579.
- ↑ Tian Na, Li Jialiang, Shi Jinming, Sui Guangchao. // International Journal of Molecular Sciences. — 2017. — Vol. 18, no. 3. — P. e191. — doi : . — .
- Atambayeva Sh. A., Khailenko V. A., Ivashchenko A. T. // Molecular Biology. — 2008. — Vol. 42, no. 2. — P. 312—320. — doi : .
- ↑ Lim Kian Huat, Ferraris L., Filloux M. E., Raphael B. J., Fairbrother W. G. // Proc. Nat. Acad. Sci. USA . — 2011. — Vol. 108, no. 27. — P. 11093—11098. — doi : . — .
- Gao Kaiping, Masuda A., Matsuura T,, Ohno K. // Nucleic Acids Research. — 2008. — Vol. 36, no. 7. — P. 2257—2267. — doi : . — .
- Clark D. . Molecular biology. — Amsterdam: Elsevier Academic Press, 2005. — 784 p. — ISBN 0-12-175551-7 .
- Warf M. B., Berglund J. A. // Trends in Biochemical Sciences. — 2010. — Vol. 35, no. 3. — P. 169—178. — doi : . — .
- Reid D. C., Chang B. L., Gunderson S. I., Alpert L., Thompson W. A., Fairbrother W. G. // RNA. — 2009. — Vol. 15, no. 12. — P. 2385—2397. — doi : . — .
- ↑ Wang Zefeng, Burge C. B. // RNA. — 2008. — Vol. 14, no. 5. — P. 802—813. — doi : . — .
- ↑ Barash Y., Calarco J. A., Gao Weijun, Pan Qun, Wang Xinchen, Shai O., Blencowe B. J., Frey B. J. // Nature . — 2010. — Vol. 465, no. 7294. — P. 53—59. — doi : . — .
- Das S., Krainer A. R. // Molecular Cancer Research. — 2014. — Vol. 12, no. 9. — P. 1195—1204. — doi : . — .
- Manley J. L., Krainer A. R. // Genes & Development. — 2010. — Vol. 24, no. 11. — P. 1073—1074. — doi : . — .
- Ke Shengdong, Zhang Xiang H.-F., Chasin L. A. // Genome Research. — 2008. — Vol. 18, no. 4. — P. 533—543. — doi : . — .
- Fairbrother W. G., Holste D., Burge C. B., Sharp P. A. // PLOS Biology . — 2004. — Vol. 2, no. 9. — P. e268. — doi : . — .
- ↑ Lynch K. W., Maniatis T. // Genes & Development. — 1996. — Vol. 10, no. 16. — P. 2089—2101. — doi : . — .
- Graveley B. R., Hertel K. J., Maniatis T. // RNA. — 2001. — Vol. 7, no. 6. — P. 806—818. — .
- Filipowicz E., Adegboyega P., Sanchez R. L., Gatalica Z. // Cancer. — 2002. — Vol. 94, no. 3. — P. 814—819. — doi : . — .
- ↑ Izquierdo J. M., Majós N., Bonnal S., Martínez C., Castelo R., Guigó R., Bilbao D., Valcárcel J. // Molecular Cell. — 2005. — Vol. 19, no. 4. — P. 475—484. — doi : . — .
- ↑ Zahler A. M., Damgaard C. K., Kjems J., Caputi M. // The Journal of Biological chemistry. — 2004. — Vol. 279, no. 11. — P. 10077—10084. — doi : . — .
- Jacquenet S., Méreau A., Bilodeau P. S., Damier L., Stoltzfus C. M., Branlant C. // The Journal of Biological Chemistry. — 2001. — Vol. 276, no. 44. — P. 40464—40475. — doi : . — .
- . // Website www.hhmi.org . Дата обращения: 26 мая 2009. 22 июня 2009 года.
- Romero P. R., Zaidi S., Fang Ya Yin, Uversky V. N., Radivojac P., Oldfield C. J., Cortese M. S., Sickmeier M., LeGall T., Obradovic Z., Dunker A. K. // Proc. Nat. Acad. Sci. USA . — 2006. — Vol. 103, no. 22. — P. 8390—8395. — doi : . — .
- Li Hong-Dong, Menon R., Omenn G. S., Guan Yuanfang. // Trends in Genetics. — 2014. — Vol. 30, no. 8. — P. 340—347. — doi : . — .
- Eksi R., Li Hong-Dong, Menon R., Wen Yuchen, Omenn G. S., Kretzler M., Guan Yuanfang. // PLOS Computational Biology . — 2013. — Vol. 9, no. 11. — P. e1003314. — doi : . — .
- Irimia M., Rukov J. L., Penny D., Roy S. W. // BMC Evolutionary Biology. — 2007. — Vol. 7. — P. 188. — doi : . — .
- Ewing B., Green P. // Nature Genetics. — 2000. — Vol. 25, no. 2. — P. 232—234. — doi : . — .
- Roest Crollius H., Jaillon O., Bernot A., Dasilva C., Bouneau L., Fischer C., Fizames C., Wincker P., Brottier P., Quétier F., Saurin W., Weissenbach J. // Nature Genetics. — 2000. — Vol. 25, no. 2. — P. 235—238. — doi : . — .
- Brett D., Pospisil H., Valcárcel J., Reich J., Bork P. // Nature Genetics. — 2002. — Vol. 30, no. 1. — P. 29—30. — doi : . — .
- Kim E., Magen A., Ast G. // Nucleic Acids Research. — 2007. — Vol. 35, no. 1. — P. 125—131. — doi : . — .
- López-Bigas N., Audit B., Ouzounis C., Parra G., Guigó R. // FEBS Letters. — 2005. — Vol. 579, no. 9. — P. 1900—1903. — doi : . — .
- Omenn G. S., Guan Yuanfang, Menon R. // Journal of Proteomics. — 2014. — Vol. 107. — P. 103—112. — doi : . — .
- Sveen A., Johannessen B., Teixeira M. R., Lothe R. A., Skotheim R. I. // BMC Genomics. — 2014. — Vol. 15. — P. 672. — doi : . — .
- Sveen A., Agesen T. H., Nesbakken A., Rognum T. O., Lothe R. A., Skotheim R. I. // Genome Medicine. — 2011. — Vol. 3, no. 5. — P. 32. — doi : . — .
- Kim E., Goren A., Ast G. // Trends in Genetics. — 2008. — Vol. 24, no. 1. — P. 7—10. — doi : . — .
- ↑ Ghigna C., Giordano S., Shen Haihong, Benvenuto F., Castiglioni F., Comoglio P. M., Green M. R., Riva S., Biamonti G. // Molecular Cell. — 2005. — Vol. 20, no. 6. — P. 881—890. — doi : . — .
- Hui Lijian, Zhang Xin, Wu Xin, Lin Zhixin, Wang Qingkang, Li Yixue, Hu Gengxi. // Oncogene. — 2004. — Vol. 23, no. 17. — P. 3013—3023. — doi : . — .
- Danckwardt S., Neu-Yilik G., Thermann R., Frede U., Hentze M. W., Kulozik A. E. // Blood. — 2002. — Vol. 99, no. 5. — P. 1811—1816. — .
- Nestler E. J. // Dialogues in Clinical Neuroscience. — 2013. — Vol. 15, no. 4. — P. 431—443. — .
- Ruffle J. K. // The American Journal of Drug and Alcohol Abuse. — 2014. — Vol. 40, no. 6. — P. 428—437. — doi : . — .
- Biliński P., Wojtyła A., Kapka-Skrzypczak L., Chwedorowicz R., Cyranka M., Studziński T. // Annals of Agricultural and Environmental Medicine. — 2012. — Vol. 19, no. 3. — P. 491—496. — .
- Olsen C. M. // Neuropharmacology. — 2011. — Vol. 61, no. 7. — P. 1109—1122. — doi : . — .
- Luco R. F., Allo M., Schor I. E., Kornblihtt A. R., Misteli T. // Cell. — 2011. — Vol. 144, no. 1. — P. 16—26. — doi : . — .
- Fairbrother W. G., Yeh R. F., Sharp P. A., Burge C. B. // Science . — 2002. — Vol. 297, no. 5583. — P. 1007—1013. — doi : . — .
- Pan Qun, Shai O., Misquitta C., Zhang Wen, Saltzman A. L., Mohammad N., Babak T., Siu H., Hughes T. R., Morris Q. D., Frey B. J., Blencowe B. J. // Molecular Cell. — 2004. — Vol. 16, no. 6. — P. 929—941. — doi : . — .
Литература
- Кребс Дж., Голдштейн Э., Килпатрик С. . Гены по Льюину. — М. : Лаборатория знаний, 2017. — 919 с. — ISBN 978-5-906828-24-8 .
Ссылки
- .
- Старокадомский, Пётр. // Сайт Biomolecula.ru (30 ноября 2007). Дата обращения: 20 марта 2018.
- 2020-06-04
- 1














