Interested Article - Позиционирование нуклеосом

- 2020-02-06
- 1
Позициони́рование нуклеосо́м — определение положения нуклеосом на последовательности ДНК эукариот. Участки ДНК могут либо входить в нуклеосомные комплексы, либо быть в составе линкерной, межнуклеосомной ДНК. Эта характеристика ДНК определяет её доступность для с белками .
Начало развития метода
Разработка методов по позиционированию нуклеосом берет своё начало в 1973 году, тогда было показано свойство ядерного хроматина под действием эндогенных нуклеаз расщепляться на серию отдельных фрагментов ДНК приблизительно равной длины. Первоначально в экспериментах в качестве нуклеазы использовалась дезоксирибонуклеаза I , впоследствии широкое распространение получила микрококковая нуклеаза .
Позиционирование нуклеосом при использовании микрококковой нуклеазы
Классический подход
Экспериментальный метод определения участков ДНК, входящих в состав нуклеосом, основан на применении микрококковой нуклеазы (метод MNase-seq). Клетки замораживают и измельчают, дают оттаять. После оттаивания содержащий хроматин гомогенат обрабатывают микроккоковой нуклеазой. Нуклеаза расщепляет линкерные, не защищённые нуклеосомами участки ДНК. При помощи протеаз и РНКаз раствор очищают от белков и РНК . ДНК экстрагируется из раствора. Применяется как колоночная хроматография , так варианты жидкостной экстракции — .
ДНК осаждается этанолом . Не все линкерные участки могли расщепиться нуклеазой. На этой стадии ДНК-продукт состоит из смеси ДНК фрагментов, соответствующих участкам, с различным покрытием нуклеосомами. Фрагменты ДНК разделяются по размеру электрофорезом на агарозном геле. ДНК экстрагируется из полоски геля, содержащей мононуклеосомный продукт. Полученные фрагменты ДНК приготавливают к секвенированию . Распространено применение высокоэффективной технологии SOLiD . В результате секвенирования получают ДНК-фрагментов. Фрагменты картируются на геном . По величине покрытия различных участков генома судят о расположении нуклеосом по последовательности ДНК .
Использование именно микроккоковой нуклеазы обусловлено тем, что она обладает как эндонуклеазной , так и экзонуклеазной активностями. Нуклеаза разрезает ДНК и последовательно отщепляет нуклеотиды со свободных концов. Недостатком микроккоковой нуклеазы является предпочтение ею, как эндонуклеазы, сайтов, составленных из чередующихся нуклеотидов dA и dT , следующими за dC или dG . В то же время она плохо распознаёт сайты из только повторяющихся dA или dT (4—6). Как экзонуклеаза микрококковая нуклеаза медленнее отщепляет нуклеотиды dC и dG. Работа фермента сильно тормозится на участках ДНК, взаимодействующих с белками. При большом времени выдержки нуклеаза начинает щепить и нуклеосомную ДНК, преимущественно в местах близости к поверхности нуклеосомы. Работа фермента в эксперименте останавливается добавлением раствора ЭДТА .
Метод MNase-seq может дополняться ультразвуковым разрушением и/или иммунопреципитацией гистона H3 (ChIP-seq), а также использованием таких ферментов, как ДНКаза (DNase-seq), транспозаза (ATAC-seq) и - (NOME-seq). Может использоваться непосредственное химическое разрушение ДНК между нуклеосомами, например, при помощи гидроксильного радикала или малых ароматических молекул вроде метидиумпропил-ЭДТА, который внедряется в двойную спираль ДНК преимущественно в участках между нуклеосомами и разрушает её в присутствии катионов Fe 2+ (метод MRE-seq) .
Позиционирование нуклеосом in vitro
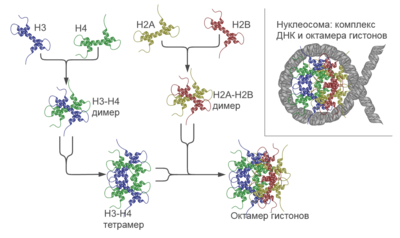
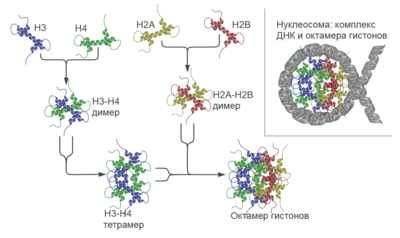
При изучении различных факторов, влияющих на распределение нуклеосом по ДНК, обычно стараются выделить компоненту влияния собственно последовательности нуклеотидов. Для этого комплекс ДНК с белками гистонами собирается in vitro из предварительно очищенной от белков ДНК и гистоновых октамеров . Количества ДНК и гистонов берутся в определённом соотношении, приблизительно один гистоновый октамер на 850 пар оснований . Далее производятся те же манипуляции, что и в обычном методе с использованием микроккоковой нуклеазы .
Предсказание сайтов связывания нуклеосом
В настоящее время развиваются методы позиционирования нуклеосом методами биоинформатики . Основа методов — экспериментально выявленные закономерности распределения нуклеосом в различных областях генома, и зависимости этих распределений от последовательности нуклеотидов ДНК . Данные по результатам экспериментального картирования нуклеосом собраны в базе данных ( англ. Nucleosome Positioning Region Database ) .
Примечания
- Hewish D. R. , Burgoyne L. A. (англ.) // Biochemical and biophysical research communications. — 1973. — Vol. 52, no. 2 . — P. 504—510. — .
- Rizzo J. M. , Sinha S. (англ.) // Methods in molecular biology (Clifton, N.J.). — 2014. — Vol. 1195. — P. 49—59. — doi : . — .
- ↑ Valouev A. , Johnson S. M. , Boyd S. D. , Smith C. L. , Fire A. Z. , Sidow A. (англ.) // Nature. — 2011. — Vol. 474, no. 7352 . — P. 516—520. — doi : . — .
- Valouev A. , Ichikawa J. , Tonthat T. , Stuart J. , Ranade S. , Peckham H. , Zeng K. , Malek J. A. , Costa G. , McKernan K. , Sidow A. , Fire A. , Johnson S. M. (англ.) // Genome research. — 2008. — Vol. 18, no. 7 . — P. 1051—1063. — doi : . — .
- Clark D. J. (англ.) // Journal of biomolecular structure & dynamics. — 2010. — Vol. 27, no. 6 . — P. 781—793. — doi : . — .
- Teif V. B. (англ.) // Briefings in bioinformatics. — 2015. — doi : . — .
- Xi L. , Fondufe-Mittendorf Y. , Xia L. , Flatow J. , Widom J. , Wang J. P. (англ.) // BMC bioinformatics. — 2010. — Vol. 11. — P. 346. — doi : . — .
- Levitsky V. G. , Katokhin A. V. , Podkolodnaya O. A. , Furman D. P. , Kolchanov N. A. (англ.) // Nucleic acids research. — 2005. — Vol. 33. — P. D67–70. — doi : . — .
Ссылки
- .
- 2020-02-06
- 1