Interested Article - Определение конформации хромосом

- 2020-08-29
- 1
Определе́ние конформа́ции хромосо́м , часто сокращённо называемые 3C ( англ. chromosome conformation capture , 3C ) — набор методов молекулярной биологии , используемых для изучения пространственной организации хроматина в ядре клетки. Их применяют для количественной оценки взаимодействий между геномными локусами , расположенными рядом в трёхмерном пространстве .
Такие взаимодействия могут возникать как следствие биологических функций, например, между промотором и энхансером или в результате случайного образования петель полимера , когда физическое движение хроматина вызывает «столкновение» локусов . При этом регуляторные элементы могут располагаться на расстоянии нескольких миллионов пар оснований от генов, которые они контролируют . Несмотря на то, что располагается на расстоянии нескольких десятков килобаз от генов, сложная конформация участка ДНК между ними позволяет им непосредственно взаимодействовать друг с другом, благодаря чему контролируется экспрессия генов .
Частоты взаимодействий могут быть проанализированы напрямую или преобразованы в расстояния и использованы для реконструкции трёхмерных структур .
Основные различия методов на основе 3C — это их возможности и область применения . Глубокое секвенирование материала, полученного с помощью 3C, также позволяет составить полногеномные карты взаимодействий.
История
Исторически микроскопия была основным методом исследования ядерной организации .
В 1984 году разработал метод иммунопреципитации хроматина, принципы которого используются в ряде методов 3C. В 2002 году Джоб Деккер предложил идею использования матрицы плотности частот взаимодействия между локусами для определения пространственной организации геномов. Эта идея послужила основой для разработки метода 3C, первая статья о котором опубликована в 2002 году Джобом Деккером и профессором в Гарвардском университете . Затем в 2006 году Марике Симонис разработала метод 4C , а Жози Дости — 5C . Далее развиваются такие методы, как ChIP-seq , Hi-C и ChIA-pet .
Экспериментальные методы
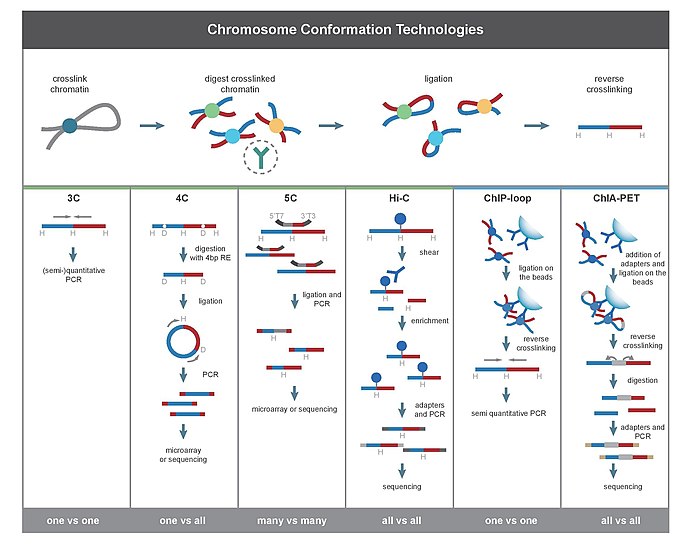
Все методы фиксации конформации хромосом начинаются с аналогичного набора манипуляций на начальном этапе, выполняемого на образце клеток. Ниже приведены основные этапы классического метода 3C.
- Обработка формальдегидом , который находящиеся рядом в пространстве участки генома и белки, таким образом замораживая взаимодействия между локусами. Чаще всего используется 1—3 % формальдегид для фиксации в течение 10-30 минут при комнатной температуре . Время реакции и концентрацию ограничивают, поскольку избыточное количество сшивок может помешать рестрикции на следующем этапе.
- Фрагментация с помощью эндонуклеаз рестрикции ( рестриктаз ). Итоговое разрешение определяется размером фрагментов. Для этой цели используют среднещепящие рестриктазы, которые распознают и режут 6 п. н. Например, или : они разрезают геном раз в каждые 4000 п. н. Таким образом, при использовании среднещепящей рестриктазы из генома человека возможно получить около 1 миллиона различных фрагментов .
- Случайное лигирование , которое производится при низких концентрациях ДНК с использованием Т4 ДНК-лигазы . В результате соединяются концы тех участков ДНК, которые были сшиты формальдегидом. Низкие концентрации обеспечивают специфичность лигирования (только между сшитыми взаимодействующими фрагментами). Впоследствии взаимодействующие локусы количественно оценивают путем амплификации лигированных фрагментов с помощью ПЦР .
- Создание парной 3С библиотеки . Термическая обработка приводит к разрыву связей и образованию линейных химерных фрагментов ДНК. В результате будет создана библиотека взаимодействующих фрагментов ДНК (3C-библиотека) .
- Real-time ПЦР позволяет оценивать вероятность взаимодействия двух конкретных участков генома. Производят подбор праймеров таким образом, что каждый праймер комплементарен своему, соответствующему локусу. В случае взаимодействия происходит отжиг обоих праймеров и амплификация фрагмента .
Базовые методы

3C
Метод захвата конформации хромосомы ( англ. chromosome conformation capture , 3C ) необходим для количественного определения взаимодействия между выбранной парой геномных локусов. Например, 3C можно использовать для изучения потенциального взаимодействия промотор-энхансер. Лигированные фрагменты детектируют с помощью ПЦР, используя праймеры к известным последовательностям .
4C
Метод замкнутого захвата конформации хромосомы ( англ. circularized chromosome conformation capture , 4С ) охватывает взаимодействия между одним выбранным локусом и остальными геномными локусами. Он используется для того, чтобы найти участок генома, который взаимодействует с данной последовательностью ДНК и представляет собой комбинацию стандартного метода 3С с .
Первые 4 этапа совпадают с этапами метода фиксации конформации хромосом. Далее последовательно проводятся фрагментация полученной 3С библиотеки рестриктазами; лигирование для циклизации фрагментов ДНК, в результате получая библиотеку «кольцевых» химерных ДНК (4C библиотека) . Инвертированная ПЦР позволяет амплифицировать неизвестную последовательность, используя известную последовательность, лигированную с ней . Анализ 4С библиотеки проводится с использованием ДНК-микрочипов .
В отличие от 3C и 5C, методы 4C не требуют предварительного знания нуклеотидных последовательностей обеих взаимодействующих хромосомных областей . Результаты, полученные с помощью 4С, являются высоко воспроизводимыми для большинства взаимодействий между проксимальными областями. На одном микрочипе можно проанализировать около миллиона взаимодействий .
5C
5C детектирует взаимодействия между всеми фрагментами в заданном регионе, причём размер этого региона обычно не превышает мегабазы. Он позволяет искать участки ДНК, которые взаимодействуют с несколькими выбранными участками генома и представляет собой комбинацию метода 3С и мультиплексной ПЦР .
Первые 4 этапа совпадают с этапами метода фиксации конформации хромосом. Далее последовательно проводятся лигирование адаптеров ко всем фрагментам с использованием Taq-лигазы; анализ 5С библиотеки проводится с использованием ДНК-микрочипов и секвенирования . 5С полезен для изучения сложных взаимодействий, однако, имеет относительно низкое покрытие. Метод не подходит для изучения комплексных взаимодействий по всему геному, поскольку для этого потребуются миллионы праймеров 5C .
Hi-C
Метод Hi-C содержит дополнительные шаги, нацеленные на обогащение итоговой библиотеки фрагментами, несущими информацию о контактах ДНК в клетке, то есть химерными фрагментами. После рестрикции производится биотинилирование концов ДНК, затем лигирование, а затем удаляются биотинилированные нуклеотиды с концов молекул ДНК. В результате биотин содержат только те фрагменты ДНК, которые подверглись всем предшествующим реакциям. Затем такие молекулы отделяются от тех, которые статистически не вступили в одну из реакций, при помощи специфичного связывания биотина стрептавидином . После приготовления библиотеки результирующих фрагментов применяют высокопроизводительное секвенирование для определения нуклеотидной последовательности химерных фрагментов . Последовательности, входящие в состав этих молекул, независимо картируется на геном, что позволяет определить, и каких участков генома они происходят, и, соответственно, какие участки генома взаимодействовали в клетке. Таким образом, проверяются все возможные парные взаимодействия между участками генома .
Учёные пытаются установить границы применимости метода Hi-C на примере исследования, посвященного скринингу первичных опухолей головного мозга . До онкоскрининга Hi-C в основном использовался для работ на клеточных линиях .
Специальные методы
К специальным методам можно отнести методы на основе захвата последовательности, single-cell методы и методы на основе иммунопреципитации. Например, single-cell Hi-C может быть использован для изучения взаимодействий в отдельных клетках .
Методы на основе захвата последовательности использует фиксацию олигонуклеотидов для обогащения библиотек 3C и Hi-C изучаемых локусов . К ним относятся: Capture-C , NG Capture-C , Capture-3C и Capture Hi-C . Эти методы позволяют достигнуть более высокого разрешения и чувствительности, чем методы на основе 4C .
Методы на основе иммунопреципитации позволяют выделить локусы, которые взаимодействуют с помощью специфических белков, например, транскрипционных факторов или инсуляторных белков . Среди них можно выделить такие методы, как ChIP-loop и ChIA-PET. ChIP-loop комбинирует 3C с ChIP-seq для детекции взаимодействия между двумя интересующими локусами, опосредованного изучаемым белком . ChIA-PET сочетает Hi-C и ChIP-seq для детекции всех взаимодействий, опосредованных изучаемым белком .
Биологическое значение
Методы 3C способствовали большому числу важных биологических открытий, включая новые данные о структурных особенностях хромосом, классификацию хроматиновых петель, а также помогли углубить знания о механизмах регуляции транскрипции (чьё нарушение может приводить к целому ряду заболеваний) .
Методы захвата конформации хромосом продемонстрировали важность пространственной близости регуляторных элементов генов. Так, например, в тканях, экспрессирующих гены глобина, контрольный участок локуса β-глобина формирует петлю вместе с данными генами. При этом петля отсутствует в тканях, где этот ген не экспрессируется .
Фиксация конформации хромосом позволила обнаружить крупный уровень их организации — так называемые ТАДы ( ), коррелирующие с изменениями в эпигенетических маркерах. Некоторые ТАДы не проявляют транскрипционной активности, в то время как активность других подавляется . Большое количество ТАДов обнаружено у D. melanogaster , мыши и человека . Основную роль в определении взаимодействий между ТАДами, энхансерами и промоторами играют транскрипционный фактор и белковый комплекс когезин . Результаты 3C экспериментов говорят о важности ориентации («лицом к лицу») связывающих мотивов CTCF и энхансер-промоторной петли. Это необходимо для корректного позиционирования энхансера относительно своей мишени .
Заболевания человека
Существует ряд болезней, вызываемых дефектами промотор-энхансерного взаимодействия . К ним относится такое заболевание крови, как , возникающее вследствие делеции энхансерного элемента ЛКО . Мутация в SBE2 энхансере, которая, в свою очередь, ослабляет экспрессию гена SHH , приводит к развитию . При этом нарушается формирование конечного мозга, разделённого на полушария. Другим примером связанных с изменением экспрессии SHH заболеваний является полидактилия второго типа PPD2 (трёхфаланговый большой палец). Она возникает из-за мутации регуляторного элемента ZRS, влияющего на усиленную продукцию SHH . Расстройство взаимодействий между промотором и энхансером влияет не только на пороки развития, но также может служить причиной онкологических заболеваний. Так, аденокарцинома лёгкого может развиваться вследствие дупликации энхансерного элемента гена MYC . T-клеточный острый лимфобластный лейкоз может быть вызван появлением нового энхансера из-за мутации в последовательности интрона .
Анализ данных

Данные, получающиеся в результате различных 3C экспериментов, характеризуются отличающимися структурой и статистическими свойствами. Поэтому для обработки каждого типа экспериментов существует свой программный пакет .
Данные Hi-C часто используются в анализе уровней полногеномной организации хроматина. В результате обработки существующими алгоритмами выделяются ТАДы, протяжённые линейные участки генома, которые связаны пространственно .
Hi-C и его производные постоянно совершенствуются. Fit-Hi-C — это метод, основанный на принципе дискретного биннинга данных. Возможны его модификации с учётом расстояния взаимодействия (уточнение начального сплайна или spline-1) и уточнением нулевой модели (spline-2). Результатом Fit-Hi-C является список попарных интрахромосомных взаимодействий с соответствующими значениями p-value и q-value .
3D организация генома может быть установлена с использованием методов спектрального разложения матрицы контактов. Каждый собственный вектор соответствует набору локусов с общими структурными свойствами (эти локусы необязательно должны быть расположены линейно друг за другом) .
Одним из искажающих факторов для технологии 3C являются частые неспецифические взаимодействия между локусами, появляющиеся в результате случайного поведения полимера. Специфичность взаимодействия между двумя локусами обязательно должна быть подтверждена на соответствующем уровне статистической значимости .
Нормализация карты контактов Hi-C
Существует два основных пути нормализации первичных данных тепловой карты контактов Hi-C. Первый — предположение о равной доступности, что означает одинаковые шансы для каждой позиции в хромосоме участвовать во взаимодействии. Cоответственно, истинный сигнал карты контактов Hi-C должен представлять собой уравновешенную матрицу (уравновешенной матрицей считается такая, для которой суммы значений по строкам и столбцам равны). Примером такого алгоритма является алгоритм , который приводит предварительную карту контактов к виду уравновешенной матрицы .
Другой способ использует предположение о том, что с каждой хромосомной позицией связана некоторая смещённость. Значение карты контактов для каждой координаты будет равняться истинному сигналу для данной позиции, умноженному на смещения для двух соседних позиций. К алгоритмам, использующим модель со смещением, относится алгоритм итеративной коррекции. В процессе его исполнения смещённость по строкам и столбцами итеративно исключается из первичной карты контактов .
Анализ мотивов ДНК
ДНК- мотивы — специфические короткие последовательности, часто длиной 8-20 нуклеотидов , которые статистически перепредставлены в наборе последовательностей с общей биологической функцией. На данный момент регуляторные мотивы дальних взаимодействий хроматина ещё не достаточно изучены .
Анализ раковых геномов
Техники, основанные на методах 3C, могут пролить свет на хромосомные перестройки в раковых геномах . Более того, они способны показывать изменения в пространственной близости регуляторных элементов и их генов-мишеней, позволяя углубить понимание структурно-функциональной организации генома в целом .
См. также
Примечания
- ↑ de Wit E. , de Laat W. (англ.) // Genes & Development. — 2012. — 1 January ( vol. 26 , no. 1 ). — P. 11—24 . — ISSN . — doi : .
- ↑ Hakim Ofir , Misteli Tom. (англ.) // Cell. — 2012. — March ( vol. 148 , no. 5 ). — P. 1068—1068.e2 . — ISSN . — doi : .
- ↑ Ay F. , Bailey T. L. , Noble W. S. (англ.) // Genome Research. — 2014. — 5 February ( vol. 24 , no. 6 ). — P. 999—1011 . — ISSN . — doi : .
- Dekker Job. (англ.) // Nature Methods. — 2005. — 20 December ( vol. 3 , no. 1 ). — P. 17—21 . — ISSN . — doi : .
- Dekker Job. (англ.) // Trends in Biochemical Sciences. — 2003. — June ( vol. 28 , no. 6 ). — P. 277—280 . — ISSN . — doi : .
- ↑ Rao Suhas S.P. , Huntley Miriam H. , Durand Neva C. , Stamenova Elena K. , Bochkov Ivan D. , Robinson James T. , Sanborn Adrian L. , Machol Ido , Omer Arina D. , Lander Eric S. , Aiden Erez Lieberman. (англ.) // Cell. — 2014. — December ( vol. 159 , no. 7 ). — P. 1665—1680 . — ISSN . — doi : .
- Varoquaux N. , Ay F. , Noble W. S. , Vert J.-P. (англ.) // Bioinformatics. — 2014. — 15 June ( vol. 30 , no. 12 ). — P. i26—i33 . — ISSN . — doi : .
- ↑ Davies James O J , Oudelaar A Marieke , Higgs Douglas R , Hughes Jim R. (англ.) // Nature Methods. — 2017. — February ( vol. 14 , no. 2 ). — P. 125—134 . — ISSN . — doi : .
- ↑ Denker Annette , de Laat Wouter. (англ.) // Genes & Development. — 2016. — 15 June ( vol. 30 , no. 12 ). — P. 1357—1382 . — ISSN . — doi : .
- ↑ Dekker J. (англ.) // Science. — 2002. — 15 February ( vol. 295 , no. 5558 ). — P. 1306—1311 . — ISSN . — doi : .
- Osborne C. S. , Ewels P. A. , Young A. N. C. (англ.) // Briefings in Functional Genomics. — 2011. — 1 January ( vol. 10 , no. 1 ). — P. 11—17 . — ISSN . — doi : .
- ↑ Simonis Marieke , Klous Petra , Splinter Erik , Moshkin Yuri , Willemsen Rob , de Wit Elzo , van Steensel Bas , de Laat Wouter. (англ.) // Nature Genetics. — 2006. — 8 October ( vol. 38 , no. 11 ). — P. 1348—1354 . — ISSN . — doi : .
- ↑ Dostie J. , Richmond T. A. , Arnaout R. A. , Selzer R. R. , Lee W. L. , Honan T. A. , Rubio E. D. , Krumm A. , Lamb J. , Nusbaum C. , Green R. D. , Dekker J. (англ.) // Genome Research. — 2006. — 1 October ( vol. 16 , no. 10 ). — P. 1299—1309 . — ISSN . — doi : .
- Albert Istvan , Mavrich Travis N. , Tomsho Lynn P. , Qi Ji , Zanton Sara J. , Schuster Stephan C. , Pugh B. Franklin. (англ.) // Nature. — 2007. — March ( vol. 446 , no. 7135 ). — P. 572—576 . — ISSN . — doi : .
- ↑ Lieberman-Aiden E. , van Berkum N. L. , Williams L. , Imakaev M. , Ragoczy T. , Telling A. , Amit I. , Lajoie B. R. , Sabo P. J. , Dorschner M. O. , Sandstrom R. , Bernstein B. , Bender M. A. , Groudine M. , Gnirke A. , Stamatoyannopoulos J. , Mirny L. A. , Lander E. S. , Dekker J. (англ.) // Science. — 2009. — 8 October ( vol. 326 , no. 5950 ). — P. 289—293 . — ISSN . — doi : .
- ↑ Fullwood Melissa J. , Liu Mei Hui , Pan You Fu , Liu Jun , Xu Han , Mohamed Yusoff Bin , Orlov Yuriy L. , Velkov Stoyan , Ho Andrea , Mei Poh Huay , Chew Elaine G. Y. , Huang Phillips Yao Hui , Welboren Willem-Jan , Han Yuyuan , Ooi Hong Sain , Ariyaratne Pramila N. , Vega Vinsensius B. , Luo Yanquan , Tan Peck Yean , Choy Pei Ye , Wansa K. D. Senali Abayratna , Zhao Bing , Lim Kar Sian , Leow Shi Chi , Yow Jit Sin , Joseph Roy , Li Haixia , Desai Kartiki V. , Thomsen Jane S. , Lee Yew Kok , Karuturi R. Krishna Murthy , Herve Thoreau , Bourque Guillaume , Stunnenberg Hendrik G. , Ruan Xiaoan , Cacheux-Rataboul Valere , Sung Wing-Kin , Liu Edison T. , Wei Chia-Lin , Cheung Edwin , Ruan Yijun. (англ.) // Nature. — 2009. — November ( vol. 462 , no. 7269 ). — P. 58—64 . — ISSN . — doi : .
- Gavrilov Alexey , Eivazova Elvira , Pirozhkova Iryna , Lipinski Marc , Razin Sergey , Vassetzky Yegor. (англ.) // Chromatin Immunoprecipitation Assays. — 2009. — P. 171—188 . — ISBN 9781603274135 . — ISSN . — doi : .
- ↑ Naumova Natalia , Smith Emily M. , Zhan Ye , Dekker Job. (англ.) // Methods. — 2012. — November ( vol. 58 , no. 3 ). — P. 192—203 . — ISSN . — doi : .
- Belton Jon-Matthew , Dekker Job. (англ.) // Cold Spring Harbor Protocols. — 2015. — June ( vol. 2015 , no. 6 ). — P. pdb.prot085175 . — ISSN . — doi : .
- ↑ Gavrilov Alexey A. , Golov Arkadiy K. , Razin Sergey V. (англ.) // PLoS ONE. — 2013. — 26 March ( vol. 8 , no. 3 ). — P. e60403 . — ISSN . — doi : .
- ↑ Stadhouders Ralph , Kolovos Petros , Brouwer Rutger , Zuin Jessica , van den Heuvel Anita , Kockx Christel , Palstra Robert-Jan , Wendt Kerstin S , Grosveld Frank , van Ijcken Wilfred , Soler Eric. (англ.) // Nature Protocols. — 2013. — 14 February ( vol. 8 , no. 3 ). — P. 509—524 . — ISSN . — doi : .
- Zhao Zhihu , Tavoosidana Gholamreza , Sjölinder Mikael , Göndör Anita , Mariano Piero , Wang Sha , Kanduri Chandrasekhar , Lezcano Magda , Singh Sandhu Kuljeet , Singh Umashankar , Pant Vinod , Tiwari Vijay , Kurukuti Sreenivasulu , Ohlsson Rolf. (англ.) // Nature Genetics. — 2006. — 8 October ( vol. 38 , no. 11 ). — P. 1341—1347 . — ISSN . — doi : .
- van de Werken Harmen J G , Landan Gilad , Holwerda Sjoerd J B , Hoichman Michael , Klous Petra , Chachik Ran , Splinter Erik , Valdes-Quezada Christian , Öz Yuva , Bouwman Britta A M , Verstegen Marjon J A M , de Wit Elzo , Tanay Amos , de Laat Wouter. (англ.) // Nature Methods. — 2012. — 9 September ( vol. 9 , no. 10 ). — P. 969—972 . — ISSN . — doi : .
- ↑ Nagano Takashi , Lubling Yaniv , Stevens Tim J. , Schoenfelder Stefan , Yaffe Eitan , Dean Wendy , Laue Ernest D. , Tanay Amos , Fraser Peter. (англ.) // Nature. — 2013. — 25 September ( vol. 502 , no. 7469 ). — P. 59—64 . — ISSN . — doi : .
- ↑ Harewood Louise , Kishore Kamal , Eldridge Matthew D. , Wingett Steven , Pearson Danita , Schoenfelder Stefan , Collins V. Peter , Fraser Peter. (англ.) // Genome Biology. — 2017. — 27 June ( vol. 18 , no. 1 ). — ISSN . — doi : .
- Burton Joshua N , Adey Andrew , Patwardhan Rupali P , Qiu Ruolan , Kitzman Jacob O , Shendure Jay. (англ.) // Nature Biotechnology. — 2013. — 3 November ( vol. 31 , no. 12 ). — P. 1119—1125 . — ISSN . — doi : .
- ↑ Schmitt Anthony D. , Hu Ming , Ren Bing. (англ.) // Nature Reviews Molecular Cell Biology. — 2016. — 1 September ( vol. 17 , no. 12 ). — P. 743—755 . — ISSN . — doi : .
- Hughes Jim R , Roberts Nigel , McGowan Simon , Hay Deborah , Giannoulatou Eleni , Lynch Magnus , De Gobbi Marco , Taylor Stephen , Gibbons Richard , Higgs Douglas R. (англ.) // Nature Genetics. — 2014. — 12 January ( vol. 46 , no. 2 ). — P. 205—212 . — ISSN . — doi : .
- Davies James O J , Telenius Jelena M , McGowan Simon J , Roberts Nigel A , Taylor Stephen , Higgs Douglas R , Hughes Jim R. (англ.) // Nature Methods. — 2015. — 23 November ( vol. 13 , no. 1 ). — P. 74—80 . — ISSN . — doi : .
- the GIANT Consortium. (англ.) // Nature Genetics. — 2008. — 14 December ( vol. 41 , no. 1 ). — P. 25—34 . — ISSN . — doi : .
- Jäger Roland , Migliorini Gabriele , Henrion Marc , Kandaswamy Radhika , Speedy Helen E. , Heindl Andreas , Whiffin Nicola , Carnicer Maria J. , Broome Laura , Dryden Nicola , Nagano Takashi , Schoenfelder Stefan , Enge Martin , Yuan Yinyin , Taipale Jussi , Fraser Peter , Fletcher Olivia , Houlston Richard S. (англ.) // Nature Communications. — 2015. — 19 February ( vol. 6 , no. 1 ). — ISSN . — doi : .
- Tiwari V. K. , Baylin S. B. (англ.) // Cold Spring Harbor Protocols. — 2009. — 1 March ( vol. 2009 , no. 3 ). — P. pdb.prot5168—pdb.prot5168 . — ISSN . — doi : .
- Horike Shin-ichi , Cai Shutao , Miyano Masaru , Cheng Jan-Fang , Kohwi-Shigematsu Terumi. (англ.) // Nature Genetics. — 2004. — 19 December ( vol. 37 , no. 1 ). — P. 31—40 . — ISSN . — doi : .
- Tolhuis Bas , Palstra Robert-Jan , Splinter Erik , Grosveld Frank , de Laat Wouter. (англ.) // Molecular Cell. — 2002. — December ( vol. 10 , no. 6 ). — P. 1453—1465 . — ISSN . — doi : .
- ↑ Cavalli Giacomo , Misteli Tom. (англ.) // Nature Structural & Molecular Biology. — 2013. — March ( vol. 20 , no. 3 ). — P. 290—299 . — ISSN . — doi : .
- Dekker Job , Marti-Renom Marc A. , Mirny Leonid A. (англ.) // Nature Reviews Genetics. — 2013. — 9 May ( vol. 14 , no. 6 ). — P. 390—403 . — ISSN . — doi : .
- Guo Ya , Xu Quan , Canzio Daniele , Shou Jia , Li Jinhuan , Gorkin David U. , Jung Inkyung , Wu Haiyang , Zhai Yanan , Tang Yuanxiao , Lu Yichao , Wu Yonghu , Jia Zhilian , Li Wei , Zhang Michael Q. , Ren Bing , Krainer Adrian R. , Maniatis Tom , Wu Qiang. (англ.) // Cell. — 2015. — August ( vol. 162 , no. 4 ). — P. 900—910 . — ISSN . — doi : .
- Krijger Peter Hugo Lodewijk , de Laat Wouter. (англ.) // Nature Reviews Molecular Cell Biology. — 2016. — 9 November ( vol. 17 , no. 12 ). — P. 771—782 . — ISSN . — doi : .
- Fritsch E. F. , Lawn R. M. , Maniatis T. (англ.) // Nature. — 1979. — 14 June ( vol. 279 , no. 5714 ). — P. 598—603 . — .
- Van der Ploeg L. H. , Konings A. , Oort M. , Roos D. , Bernini L. , Flavell R. A. (англ.) // Nature. — 1980. — 14 February ( vol. 283 , no. 5748 ). — P. 637—642 . — .
- Jeong Y. (англ.) // Development. — 2006. — 15 February ( vol. 133 , no. 4 ). — P. 761—772 . — ISSN . — doi : .
- Wieczorek Dagmar , Pawlik Barbara , Li Yun , Akarsu Nurten A. , Caliebe Almuth , May Klaus J.W. , Schweiger Bernd , Vargas Fernando R. , Balci Sevim , Gillessen-Kaesbach Gabriele , Wollnik Bernd. (англ.) // Human Mutation. — 2010. — January ( vol. 31 , no. 1 ). — P. 81—89 . — ISSN . — doi : .
- Zhang Xiaoyang , Choi Peter S , Francis Joshua M , Imielinski Marcin , Watanabe Hideo , Cherniack Andrew D , Meyerson Matthew. (англ.) // Nature Genetics. — 2015. — 14 December ( vol. 48 , no. 2 ). — P. 176—182 . — ISSN . — doi : .
- Mansour M. R. , Abraham B. J. , Anders L. , Berezovskaya A. , Gutierrez A. , Durbin A. D. , Etchin J. , Lawton L. , Sallan S. E. , Silverman L. B. , Loh M. L. , Hunger S. P. , Sanda T. , Young R. A. , Look A. T. (англ.) // Science. — 2014. — 13 November ( vol. 346 , no. 6215 ). — P. 1373—1377 . — ISSN . — doi : .
- Dixon Jesse R. , Selvaraj Siddarth , Yue Feng , Kim Audrey , Li Yan , Shen Yin , Hu Ming , Liu Jun S. , Ren Bing. (англ.) // Nature. — 2012. — 11 April ( vol. 485 , no. 7398 ). — P. 376—380 . — ISSN . — doi : .
- Yardımcı Galip Gürkan , Noble William Stafford. (англ.) // Genome Biology. — 2017. — 3 February ( vol. 18 , no. 1 ). — ISSN . — doi : .
- ↑ Imakaev Maxim , Fudenberg Geoffrey , McCord Rachel Patton , Naumova Natalia , Goloborodko Anton , Lajoie Bryan R , Dekker Job , Mirny Leonid A. (англ.) // Nature Methods. — 2012. — 2 September ( vol. 9 , no. 10 ). — P. 999—1003 . — ISSN . — doi : .
- Lajoie Bryan R. , Dekker Job , Kaplan Noam. (англ.) // Methods. — 2015. — January ( vol. 72 ). — P. 65—75 . — ISSN . — doi : .
- Zambelli Federico , Pesole Graziano , Pavesi Giulio. (англ.) // Briefings in Bioinformatics. — 2012. — 19 April ( vol. 14 , no. 2 ). — P. 225—237 . — ISSN . — doi : .
- Wong Ka-Chun , Li Yue , Peng Chengbin. (англ.) // Bioinformatics. — 2015. — 26 September ( vol. 32 , no. 3 ). — P. 321—324 . — ISSN . — doi : .
- Taberlay Phillippa C. , Achinger-Kawecka Joanna , Lun Aaron T.L. , Buske Fabian A. , Sabir Kenneth , Gould Cathryn M. , Zotenko Elena , Bert Saul A. , Giles Katherine A. , Bauer Denis C. , Smyth Gordon K. , Stirzaker Clare , O'Donoghue Sean I. , Clark Susan J. (англ.) // Genome Research. — 2016. — 6 April ( vol. 26 , no. 6 ). — P. 719—731 . — ISSN . — doi : .
- 2020-08-29
- 1